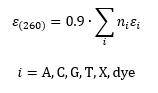Back to top
Foire aux Questions - Synthèse d'Oligonucléotides
Produits et Services
Oui, nous proposons des sondes MGB avec différents Dyes.
La modification (3' MGB-Q530) peut être commandée directement sur notre boutique en ligne.
Oui, Microsynth propose des Oligos contenant des bases dégénérées comme produit standard. Les bases dégénérées peuvent être saisies directement dans la boutique en ligne à l’aide du code IUB correspondant (voir également la boutique en ligne). Toutes les bases dégénérées (bases mixtes) sont synthétisées avec des réactifs de synthèse prémélangés de haute précision, garantissant la meilleure distribution équimolaire possible des bases dégénérées dans l’Oligo.
Non, nous vous recommandons d'utiliser Primer-BLAST pour concevoir vos amorces.
Les oligonucléotides thiol-modifiés achetés chez Microsynth sont déjà réduits à leur forme active. Vous pouvez dissoudre les oligonucléotides thiol-modifiés dans le solvant désiré et les utiliser directement. Une fois dissous, nous recommandons fortement de conserver les échantillons à -20°C ou moins.
Au fil du temps, après avoir été exposés à l'air ou à un solvant pouvant agir comme un agent oxydant, tel que le DMSO, la formation réversible d'une liaison disulfure intramoléculaire peut se produire. Dans ce cas, les oligonucléotides modifiés par des thiols doivent être manipulés avec un agent réducteur tel que le DTT ou le TCEP.
Les deux combinaisons sont possibles. Cependant, d'après notre expérience, HEX offre de meilleures performances avec BHQ-2 qu'avec BHQ-1. Par conséquent, nous recommandons d'utiliser HEX en combinaison avec BHQ-2 pour des résultats optimaux.
Questions Relatives à la Commande
Pour commander des siRNAs de contrôle, consultez notre site web ici. Les séquences listées peuvent être facilement commandées dans différentes échelles de synthèse et niveaux de purification sur la boutique en ligne de Microsynth.
Oui, nous pouvons livrer des oligos dans des plaques de 384 puits. Veuillez télécharger notre bon de commande en format excel sous la rubrique Téléchargement dans la boutique en ligne. Collez vos séquences dans le fichier et envoyez le fichier excel par e-mail à oligo.support@microsynth.ch. Ne pas envoyer la feuille excel directement dans la boutique en ligne. Nous reviendrons vers vous avec une offre et plus de détails sur votre commande.
Remplacez les différentes bases d'ADN/ARN ou d'ARN 2'-O-méthyle de votre séquence par les modifications internes 5, 6, 7 et 8 (par exemple TTAGCrAArGTTrUrC -> TTAGC5A6TT78). Connectez-vous à notre boutique en ligne et entrez votre séquence dans le champ de séquence. Définissez la modification dans le menu déroulant pour 5, 6, 7, 8 (soit des bases ARN/ADN ou 2'-O-Méthyl-ARN).
Vous pouvez déplacer la position des oligonucléotides dans la plaque de 96 puits en plaçant une position vide. Pour ce faire, entrez un jeu de données d'oligo avec les propriétés suivantes :
i. Oligoname: Leerposition
ii. Échelle : Genomics
iii. La purification : Desalted
iv. Séquence : TT
La quantité minimale d'oligos pour recevoir une plaque de 96 puits est de 40 oligonucléotides.
- Sélectionnez un nucléotide d'ADN "T" dans votre séquence et remplacez le "T" par "5".
- Sélectionnez votre Dye (par exemple FAM-dT) dans le menu déroulant sous "Modification interne (5=...)".
- Suivez les instructions suivantes et soumettez votre commande
Le screening initial en utilisant différents siRNAs : pour une telle application, nos clients utilisent généralement des siRNAs dessalés qui ont subi une purification Cartridge. On obtient ainsi des niveaux de pureté d'environ 80 %, ce qui est relativement suffisant pour un premier criblage.
Pour le criblage avancé ou les cultures cellulaires : si votre expérience devient plus sophistiquée et donc plus coûteuse, nous recommandons généralement de choisir la purification HPLC (~85%) ou même la purification PAGE (~95%).
Pour des quantités plus importantes, à partir de 5 mg, nous proposons des siRNA à faible taux d'endotoxine, spécifiquement destinés à l'expérimentation animale. Veuillez nous contacter pour plus d'informations.
En standard, il y a un groupe OH aux 5' et 3' d'un oligo.
Les coefficients d'extinction molaire des oligonucléotides sont calculés selon le "base composition model". (1-3) Ce modèle inclut l'absorption des nucléobases individuelles et les modifications potentielles des bases et/ou des Dyes. Des valeurs plus précises pour les oligonucléotides non modifiés sont obtenues à partir du "nearest-neighbor model", qui tient compte des effets d'empilement des nucléobases adjacentes. Selon nos calculs, ce modèle réduit le coefficient d'extinction à environ 90 % par rapport au modèle de composition des bases. Par conséquent, les coefficients d'extinction des oligonucléotides sont estimés selon l'équation ci-dessous.
- Tataurov A.V., You Y., and Owczarzy R. (2008) Predicting ultraviolet spectrum of single stranded and double stranded deoxyribonucleic acids, Biophys. Chem. 133, 66-70.
- Fasman, G.D. (Ed.) (1975) Handbook of Biochemistry and Molecular Biology, Volume 1: Nucleic Acids, pp 589, 3rd edition, CRC Press.
- Cavaluzzi M.J., and Borer P.N. (2004) Revised UV extinction coefficients for nucleoside-5'-monophosphates and unpaired DNA and RNA, Nucleic Acids Res. 32, e13.