
Back to top
Oligos Antisens

- 2'-O-Méthyle
- 2'-Fluoro
- MOE (2'-méthoxyéthyle)
- LNA (locked nucleic acid)
- S-cEt (éthyle contraint, sur demande)
- PTO (phosphorothioate)
- Gapmers
- GalNAc
Caractéristiques et Avantages
- Tous les ASOs sont contrôlés par MALDI-TOF MS avant l'expédition.
- Microsynth est un fournisseur ASO renommé pour les instituts de recherche et les entreprises pharmaceutiques/biotechniques.
- Oligonucléotides à faible taux d'endotoxine, veuillez demander un devis.
- Les échelles de synthèse jusqu'à 15 µmol sont expédiés dans les 4 à 6 jours
- Pour les applications in vivo, les délais de production peuvent atteindre 3 semaines, y compris les tests d'endotoxine.
- Des scientifiques formés et expérimentés sont heureux de vous aider.
- Des prix compétitifs
- Des rendements garantis élevés
- HPLC analytique disponible
- Certificat d'analyse disponible
- Portail en ligne facile à utiliser, doté d'une série d'outils utiles (par exemple, suivi et historique des commandes, option de recherche et de commande pratique)
Vue d'Ensemble
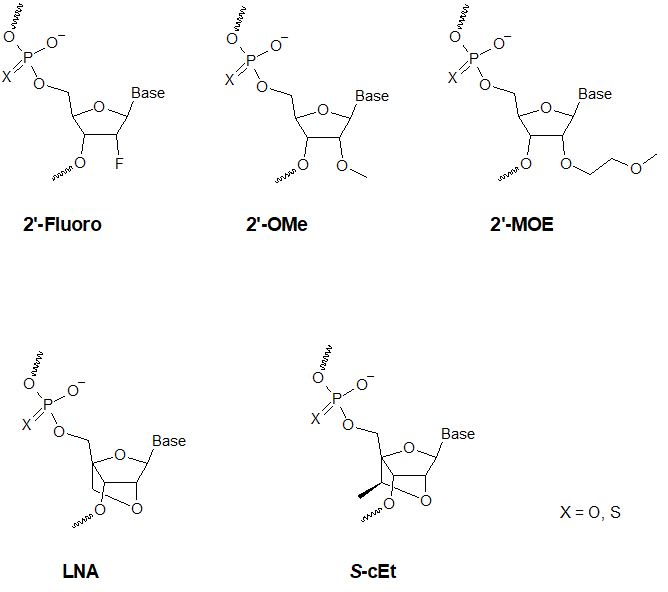
- Modifications des PTO (phosphorothioates) : Les PTO contiennent un atome de soufre à la place d'un atome d'oxygène dans la liaison inter nucléotidique de l'ADN ou de l'ARN. Cette modification du squelette normal phosphodiester est caractérisée par une augmentation de l'absorption cellulaire, une résistance élevée aux nucléases et l'activation de l'activité de RNAse H.
- Modifications 2'-O-Me-RNA : L'incorporation de nucléotides d'ARN 2'-O-Méthyle induit une résistance à une grande variété de nucléases, en particulier la RNase. En outre, les oligonucléotides 2'-OMe présentent une affinité légèrement accrue envers leur séquence cible d'ARNm complémentaire, formant ainsi des duplex hybrides plus stables par rapport à leurs homologues d'ADN ou d'ARN non modifiés. Cela permet la formation d'hybrides plus stables avec des brins d'ARN complémentaires que ce ne serait le cas pour des séquences d'ADN et d'ARN non modifiées.
- 2'– MOE-RNA modifications : Les oligonucléotides incorporant des nucléotides modifiés par le 2'-O-méthoxyéthyle (MOE), peuvent supporter la plupart, sinon la totalité des mécanismes d'action antisens. D'autres caractéristiques distinctives clés sont la résistance aux nucléases, une toxicité plus faible, une spécificité de liaison à la cible supérieure, ainsi qu'une affinité accrue envers l'ARN complémentaire. Pour des informations plus détaillées sur les oligonucléotides antisens 2'-MOE de Microsynth, veuillez consulter le flyer sur le côté droit.
-
Modifications LNA : L'ARN contenant des oligonucléotides offre une affinité sensiblement accrue pour son brin complémentaire, par rapport aux oligonucléotides traditionnels d'ADN ou d'ARN. Cette caractéristique et la résistance élevée aux nucléases des LNA qui en découle se traduisent par une sensibilité et une spécificité sans précédent et font des oligonucléotides de LNA™ des produits idéaux pour les applications antisens.
- Modifications S-cEt : les oligonucléotides cEt (nucléotides éthyl contraints) sont l'évolution des oligonucléotides LNA. Ils ont été développés pour des applications antisens et sont principalement utilisés dans les gapmers. Ces ASO présentent une stabilité supérieure à celle des LNA en ce qui concerne la dégradation des nucléases, sans compromettre la sélectivité de la liaison ou la stabilité de l'hybridation.
- N-acétylgalactosamine (GalNAc) : La conjugaison de la N-acétylgalactosamine (GalNAc) est devenue une stratégie clinique majeure pour la délivrance d'oligonucléotides aux hépatocytes (cellules du foie). Ces conjugués sont efficacement internalisés par liaison au récepteur d'asialoglycoprotéine (ASGR), qui présente une grande affinité pour les oligosaccharides terminés par la N-acétyl galactosamine.
Pour en savoir plus sur les spécifications de ces cinq types de modifications, veuillez consulter le tableau ci-dessous.
Spécifications clés :
|
Modifications |
Position |
Echelle de Synthèse [µmol] | Purification1 | |||||
|
5’ |
3’ |
Int. |
0.04 |
0.2 |
1 |
15 |
HPLC + Dialyse |
|
|
PTO |
x |
x |
x |
x |
x |
x |
||
| 2’-O-Me-RNA |
x |
x |
x |
x |
x |
x |
x |
x |
| 2'-MOE-RNA | x | x | x | x | x | x | x | x |
| LNA | x | x | x | x | x | x | x | x |
| GalNAc | x | x | x | x | x | |||
1 Nous recommandons vivement de commander des ASO avec une purification "HPLC + Dialyse". L'échange de Na+ sel est nécessaire pour éliminer les sels toxiques de la purification HPLC. Pour les tests in vivo, nous recommandons nos oligonucléotides à faible endotoxine.
Echelles de Synthèse
Entièrement 2' OMe, 2' MOE en combinaison avec ou sans PTO
Desalted
| Synthesis scale1 | Length Restriction | Guaranteed Yield2 | Production Time [wd] | |
| [OD260] | [nmol]3 | |||
| 0.04 µmol | 13 - 30 | 3 | 15 | 2-3 |
| 0.2 µmol | 10 | 50 | 2-3 | |
| 1.0 µmol | 30 | 150 | 2-3 | |
| 15 µmol | 600 | 3'000 | 3-5 | |
HPLC
| Synthesis scale1 | Length Restriction | Guaranteed Yield2 | Production Time [wd] | |
| [OD260] | [nmol]3 | |||
| 0.04 µmol | 13 - 30 | 1 | 5 | 3-4 |
| 0.2 µmol | 3 | 15 | 3-4 | |
| 1.0 µmol | 10 | 50 | 3-4 | |
| 15 µmol | 300 | 1'500 | 4-6 | |
HPLC & Dialysis
| Synthesis scale1 | Length Restriction | Guaranteed Yield2 | Production Time [wd] | |
| [OD260] | [nmol]3 | |||
| 0.04 µmol | 13 - 30 | - | - | - |
| 0.2 µmol | 3 | 15 | 3-5 | |
| 1.0 µmol | 10 | 50 | 3-5 | |
| 15 µmol | 250 | 1'250 | 5-7 | |
PAGE
| Synthesis scale1 | Length Restriction | Guaranteed Yield2 | Production Time [wd] | |
| [OD260] | [nmol]3 | |||
| 0.04 µmol | 13 - 30 | 0.5 | 2.5 | 3-4 |
| 0.2 µmol | 2 | 10 | 3-4 | |
| 1.0 µmol | 7 | 35 | 3-4 | |
| 15 µmol | - | - | - | |
Gapmers (y compris ADN et 2' OMe ou 2' MOE)
HPLC
| Synthesis scale1 | Length Restriction | Guaranteed Yield2 | Production Time [wd] | |
| [OD260] | [nmol]3 | |||
| 0.04 µmol | 13 - 30 | 1 | 5 | 3-4 |
| 0.2 µmol | 3 | 15 | 3-4 | |
| 1.0 µmol | 8 | 40 | 3-4 | |
| 15 µmol | 300 | 1'500 | 4-6 | |
HPLC & Dialysis
| Synthesis scale1 | Length Restriction | Guaranteed Yield2 | Production Time [wd] | |
| [OD260] | [nmol]3 | |||
| 0.04 µmol | 13 - 30 | - | - | - |
| 0.2 µmol | 3 | 15 | 3-5 | |
| 1.0 µmol | 8 | 40 | 3-5 | |
| 15 µmol | 250 | 1'250 | 5-7 | |
PAGE
| Synthesis scale1 | Length Restriction | Guaranteed Yield2 | Production Time [wd] | |
| [OD260] | [nmol]3 | |||
| 0.04 µmol | 13 - 30 | 0.5 | 2.5 | 3-4 |
| 0.2 µmol | 2 | 10 | 3-4 | |
| 1.0 µmol | 7 | 35 | 3-4 | |
| 15 µmol | - | - | - | |
Gapmers (y compris ADN et LNA)
HPLC
| Synthesis scale1 | Length Restriction | Guaranteed Yield2 | Production Time [wd] | |
| [OD260] | [nmol]3 | |||
| 0.04 µmol | 13 - 30 | 1 | 5 | 3-4 |
| 0.2 µmol | 2 | 10 | 3-4 | |
| 1.0 µmol | 6 | 30 | 3-4 | |
| 15 µmol | 250 | 1'250 | 4-6 | |
HPLC & Dialysis
| Synthesis scale1 | Length Restriction | Guaranteed Yield2 | Production Time [wd] | |
| [OD260] | [nmol]3 | |||
| 0.04 µmol | 13 - 30 | - | - | - |
| 0.2 µmol | 2 | 10 | 3-5 | |
| 1.0 µmol | 6 | 30 | 3-5 | |
| 15 µmol | 200 | 1'000 | 5-7 | |
PAGE
| Synthesis scale1 | Length Restriction | Guaranteed Yield2 | Production Time [wd] | |
| [OD260] | [nmol]3 | |||
| 0.04 µmol | 13 - 30 | 0.5 | 2.5 | 3-4 |
| 0.2 µmol | 2 | 10 | 3-4 | |
| 1.0 µmol | 5 | 25 | 3-4 | |
| 15 µmol | - | - | - | |
1 L'échelle de synthèse représente la quantité initiale de bases 3' (matériel de départ).
2 Nos rendements garantis et moyens sont mesurés en DO.
3 Les rendements indiqués en nmol représentent un exemple de calcul pour un 20mer. Pour ce calcul, la règle empirique suivante a été appliquée : nmol d'oligo = DO x 100/longueur d'oligo. Veuillez noter que ce calcul est basé sur des séquences ayant une distribution pratiquement homogène des 4 bases d'ADN ; il peut varier pour des séquences ayant une teneur élevée en GC >70 %, etc.
Comment Commander
- Entrez dans notre boutique en ligne
- Cliquez sur DNA dans le domaine bleu "DNA/RNA Synthesis"
- Sélectionnez soit Normal Entry pour ecrire ou copier/coller les informations de la séquence souhaitée, etc., soit Upload Entry en utilisant notre template Excel pratique (téléchargeable sur notre boutique en ligne).
- Entrez 5,6,7,8 pour chaque base distincte à remplacer dans la séquence
- Définir la modification interne (5=...) en sélectionnant la modification souhaitée dans le menu déroulant (par exemple 5=...2'-O-Méthyl-ARN-A, 6=...2'-O-Méthyl-ARN-C etc.)
- Entrez 5,6,7,8 pour chaque base distincte à remplacer dans la séquence
- Définir la modification interne (5=...) en sélectionnant la modification souhaitée dans le menu déroulant (par exemple 5=...2'-Méthoxyéthyl-ribo-A, 6=...2'-Méthoxyéthyl-ribo-mC etc.)
- Entrez 5,6,7,8 pour chaque base distincte à remplacer dans la séquence
- Définir la modification interne (5=...) en sélectionnant la modification souhaitée dans le menu déroulant (par exemple 5=...LNA-A, 6=...LNA-C etc.)
- cEt ASO sont disponibles sur demande uniquement. Les instructions de commande sont jointes à l'offre.
- Les ASO à faible endotoxine sont disponibles sur demande uniquement. Les instructions de commande sont jointes à l'offre.

