Back to top
Analyse Métagénomique du Génome Entier

- Étudier le potentiel génétique des échantillons de votre communauté bactérienne
- Générer une analyse taxonomique sans hypothèse
Aperçu
Considérations avant de lancer un projet de métagénomique shotgun :
- Quantités d'échantillons et origine ?
- Profondeur de séquençage (sensibilité) ?
- Focus sur le potentiel génétique ou sur la taxonomie ?
- Complexité et composition de la communauté ?
- Étude descriptive et/ou empirique ?
Laissez-nous vous guider - de la conception à l'analyse
Exemples de projets utilisant la métagénomique shotgun :
- Analyse des communautés microbiennes
- Changement des communautés et du potentiel génomique en fonction de l'évolution des conditions environnementales
- Adaptations évolutives dans les micro-environnements
- Analyse au niveau des gènes des communautés microbiennes non cultivables
- Détection de virus dans les tissus et les exsudats de mammifères
Applications liées à la métagénomique shotgun :
- Métagénomique des amplicons
- Métatranscriptomique shotgun
- Reséquençage des bactéries
Déroulement
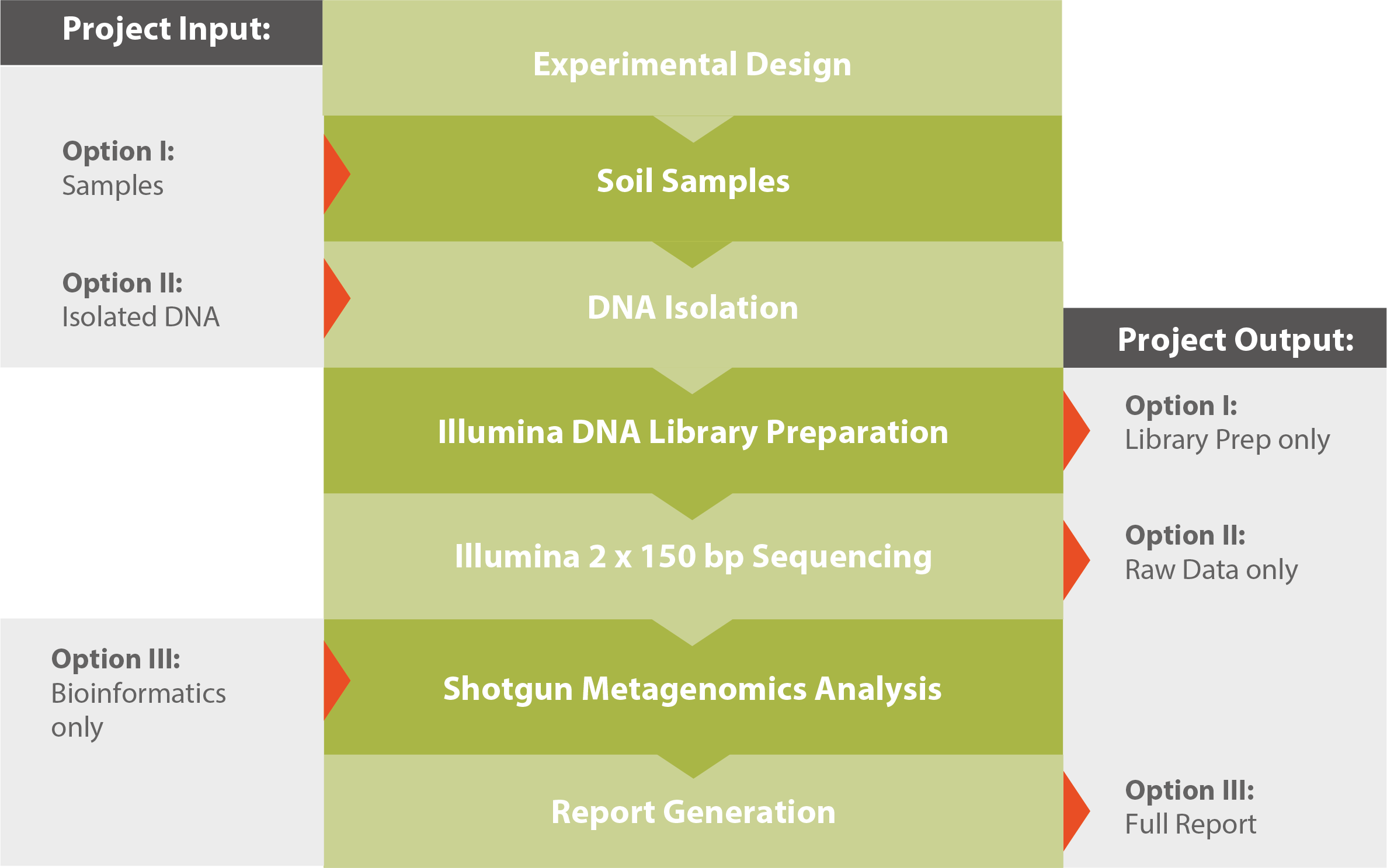
Pour plus d'informations et une description technique détaillée, veuillez télécharger notre note d'application "Shotgun Metagenomics" (voir les téléchargements correspondants).
Résultats
Contrairement aux études de métagénomique basées sur les amplicons, la métagénomique shotgun n'est pas basée sur un seul gène marqueur phylogénétique mais prend en compte tout l'ADN prélevé sur un échantillon. Ainsi, les organismes eucaryotes et procaryotes d'une communauté microbienne peuvent être analysés simultanément. La métagénomique n'est pas limitée à la composition taxonomique d'une communauté mais révèle également l'inventaire des gènes fonctionnels.
L'analyse des jeux de données de métagénomique shotgun est difficile en raison de leur taille. L'alignement des reads sur une base de données de référence est le principal facteur limitant et nécessite des ressources matérielles et logicielles adéquates. Notre module d'analyse de métagénomique shotgun fonctionne sur des serveurs de haute performance en utilisant les outils les plus rapides disponibles. Le module vous aidera à répondre aux questions suivantes :
- Quelle est la composition taxonomique (eucaryotes et procaryotes) de la communauté microbienne ? (voir figure 1)
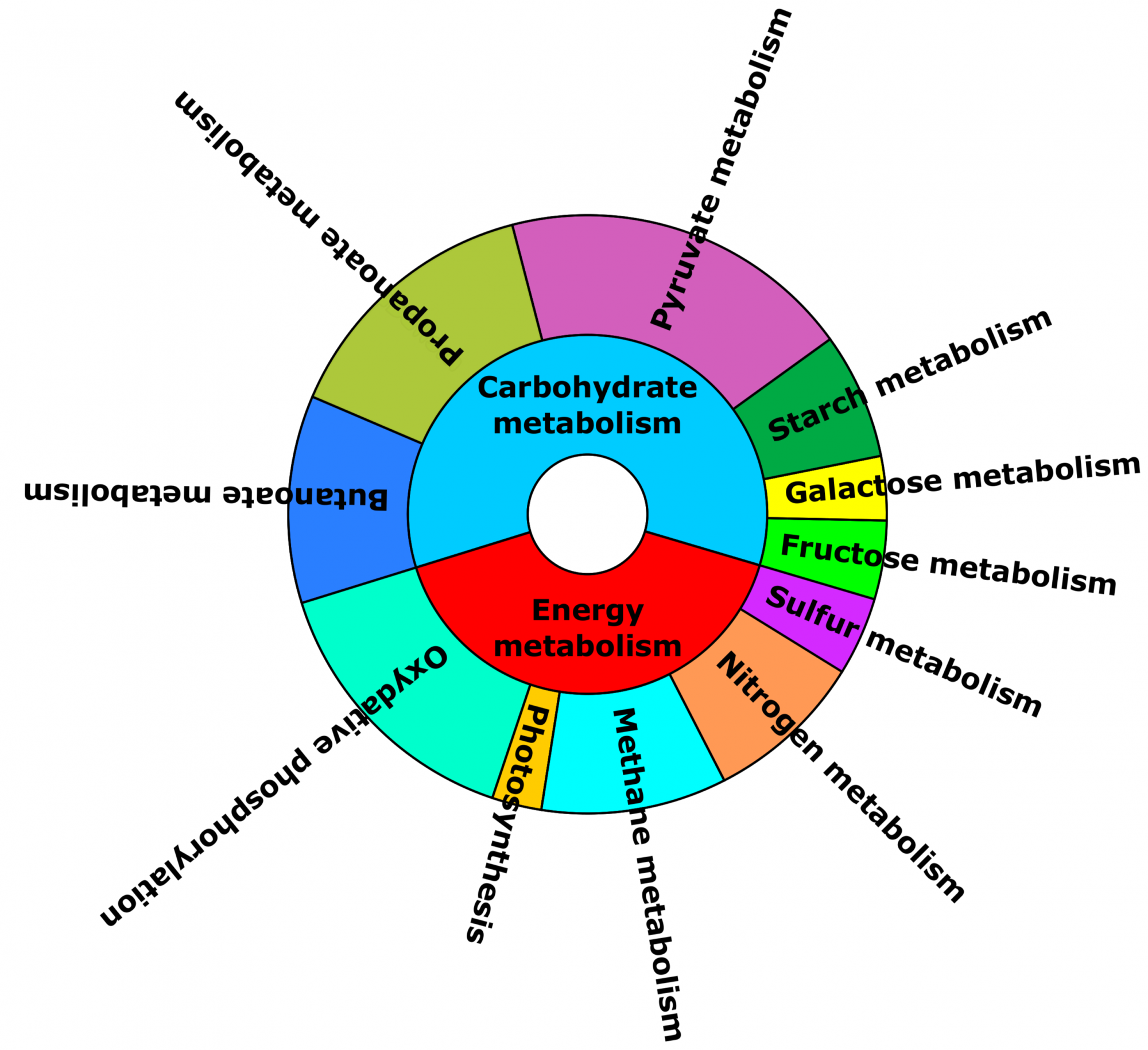
- Quel est le potentiel fonctionnel de la communauté microbienne ? (voir figure 2)
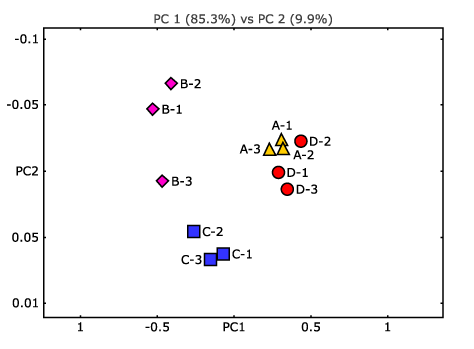
- Existe-t-il des caractéristiques taxonomiques et fonctionnelles différentes entre les communautés ou les conditions ? (voir figure 3)

Figure 1: Nuage de mots représentant la composition bactérienne d'une communauté au niveau du phylum. La taille des mots est proportionnelle à l'abondance de l'embranchement.
Délai d'exécution
- Livraison des données dans les 25 jours ouvrables suivant la réception de l'échantillon (y compris la préparation de la bibliothèque et le séquençage)
- 10 jours ouvrables supplémentaires pour l'analyse des données (bioinformatique)
- Service express possible sur demande