Back to top
nanoCORE Amplicon Metagenomics (16S/18S/ITS)

Explorez les communautés microbiennes avec le service nanoCORE Amplicon Metagenomics de Microsynth, basé sur le séquençage Oxford Nanopore. Offrant un séquençage de pleine longueur basé sur des marqueurs, notre service fournit une résolution taxonomique inégalée jusqu'au niveau des espèces. Que vous analysiez des échantillons environnementaux, suiviez les changements dans les sols agricoles ou étudiiez la diversité microbienne dans des contextes médicaux, notre service précis et efficace soutient vos recherches avec une précision maximale.
Caractéristiques et Avantages
Rapide
- Résultats livrés sous 3 semaines après réception des échantillons.
Pratique
- Forfait complet : de l'isolement de l'échantillon à la préparation avancée de la bibliothèque, au séquençage et à l'analyse bioinformatique complète.
- Utilisez notre boîte de dépôt à proximité pour l'envoi gratuit des échantillons.
- Commencez votre analyse avec un seul échantillon !
Économique
- Une méthode économique pour explorer votre communauté microbienne.
- Choisissez les étapes de votre analyse et nous vous proposerons des tarifs fixes très compétitifs, adaptés à vos besoins spécifiques.
Flexibilité accrue
- Aucune restriction de taille pour le locus marqueur.
- Séquençage des polymorphismes de longueur.
- Résolution taxonomique améliorée.
Workflow
Le flux de travail pour le service nanoCORE Amplicon Metagenomics est illustré dans le graphique ci-dessous. Veuillez noter que notre processus modulaire vous offre différentes options d'entrée.
Résultats
Analyse Bioinformatique Standard
Avec le module d'analyse bioinformatique standard de Microsynth pour nanoCORE Amplicon Metagenomics, vous recevrez des informations complètes :
- Rapport détaillé (au format interactif .html)
Ce rapport détaillé vous guide à travers les données et permet de trier et filtrer les résultats clés de manière interactive. - Operational Taxonomic Units (OTUs)
Les OTUs au format .fasta. - Abondance des OTUs, Taxonomie et Confiance des Prédictions (au format .tsv)
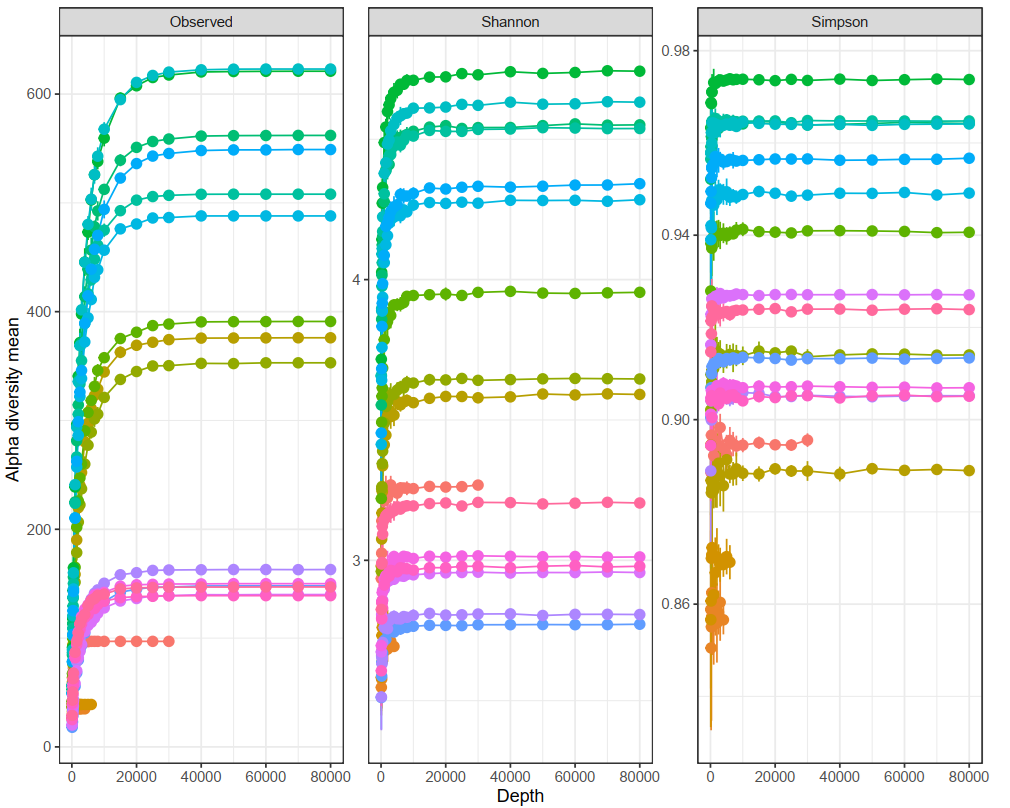
Informations sur l'abondance des OTUs, leur taxonomie et le degré de confiance des prédictions. - Analyse de la Diversité Intra- et Interéchantillon et Courbes de Rarefaction (au format .tsv et .pdf, voir Figure 1 et 2)
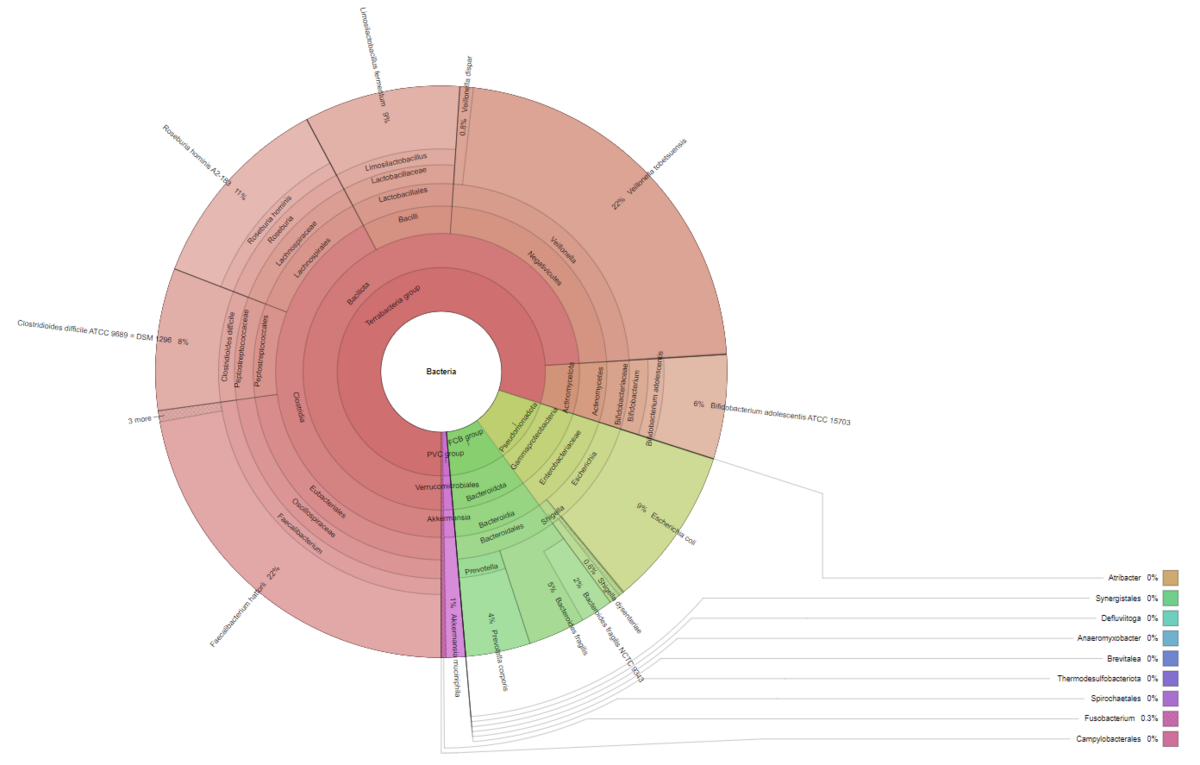
Explorez la diversité au sein des échantillons et estimez la couverture métagénomique à l'aide de courbes de rarefaction. - Visualisation du Métagénome via des Diagrammes Krona (au format interactif .html, voir Figure 3)
Interprétez et explorez facilement vos données métagénomiques grâce à des diagrammes Krona interactifs. - Analyse en Composantes Principales (au format .tsv et .pdf)
Visualisez et interprétez des relations complexes grâce à l'analyse en composantes principales. - Visualisation de l'Analyse Statistique
Accédez à des représentations visuellement informatives des analyses statistiques.
Ces résultats fournissent une compréhension complète de votre expérience, depuis une vue d'ensemble jusqu'au niveau nucléotidique, vous permettant de prendre des décisions éclairées et de tirer des conclusions pertinentes.
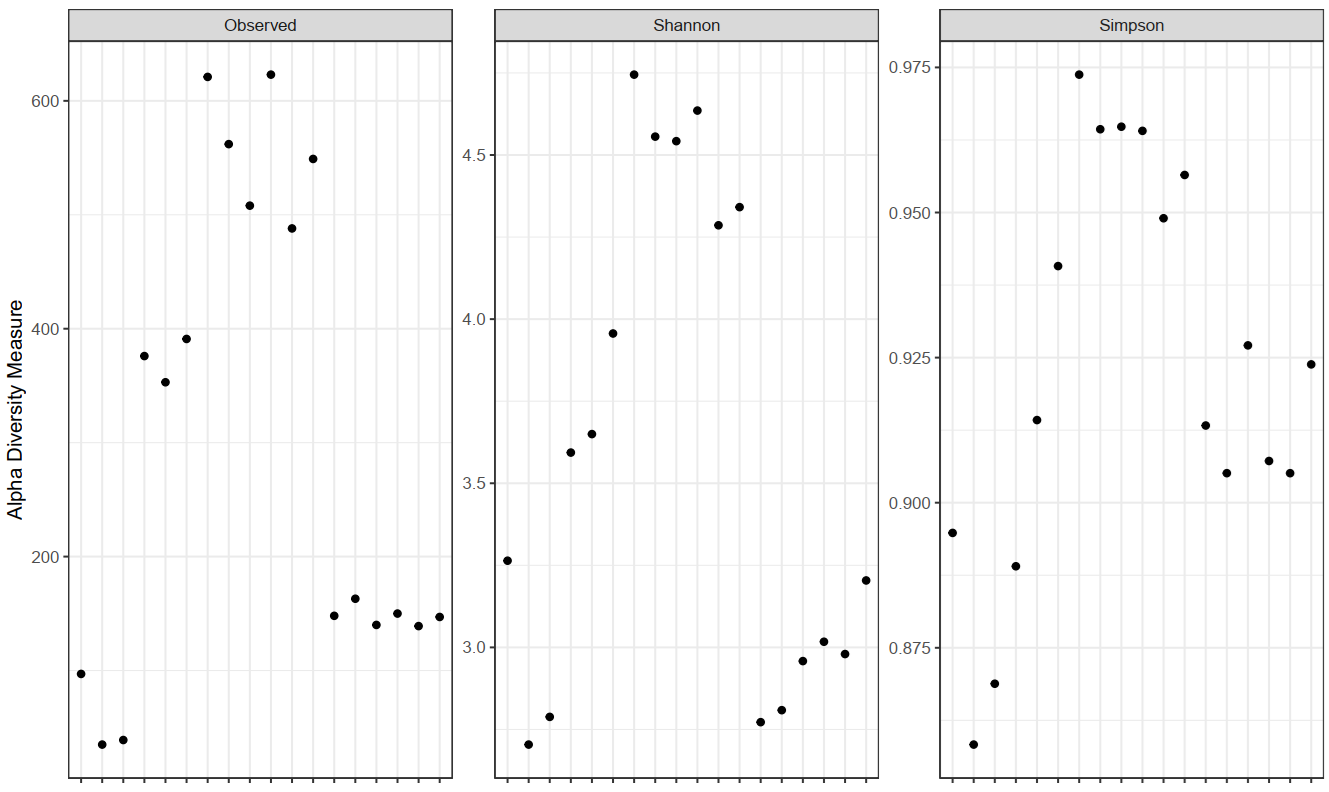
Figure 1 : Mesures de la diversité alpha pour la communauté analysée, y compris la richesse observée, les indices de Simpson et les indices de diversité de Shannon représentant la richesse et la régularité estimées.
Délai d'exécution
- Livraison des données dans les 10 jours ouvrables suivant la réception de l'échantillon (y compris la préparation de la library, le séquençage et l'analyse des données/la bio-informatique).
- 5 jours supplémentaires sont nécessaires pour l'isolement de l'ADN.