Back to top
miCORE mRNA Sequencing

- die unter unterschiedlichen Bedingungen erhaltenen Genexpressionsprofile zu vergleichen
- Genetische Profile von der Transkript- bis zur Ebene der Signalwege zu untersuchen
Übersicht
Überlegungen vor dem Start eines mRNA-Sequenzierungsprojekts:
- Wissenschaftliches Ziel
- Ausgangsmaterial & gewünschte Dienstleistungen (Isolierung bis Bioinformatik)
- Organismus und RNA-Fraktion (Poly(A)-Anreicherung, Ribo-Abreicherung)
- Verfügbare Daten (Modellorganismus, Referenzgenom)
- Sequenzierungstiefe (Empfindlichkeit)
- Replikate (Sicherheit)
- Sequenzierungslänge (Spezifität)
- Probenmengen (Komplexität)
- Versuchsaufbau (Anzahl der Proben, Wiederholungen, zu vergleichende Bedingungen)
Lassen Sie sich von uns beraten - vom Design bis zur Analyse
Beispielprojekte mit mRNA-Sequenzierung:
- Funktionelle Protein- und Signalwegstudien
- Krankheitsbedingte Veränderungen der Genexpression
- Knock-out oder Knock-in Experimente
- Teil einer Omics-Charakterisierung
- Funktionelle Veränderungen aufgrund von Spezies-Interaktionen
- Entdeckung von neuen Genen oder nicht-kodierender regulatorischer RNA
- Erkennung von RNA-Varianten
- Medikamenten-Tests
Anwendungen im Zusammenhang mit der mRNA-Sequenzierung:
- Shotgun metatranscriptomics
- Small RNA sequencing
Workflow
Resultate
Ohne Bioinformatik
Rohdaten
Wenn kein bioinformatisches Analysemodul bestellt wird, liefert Microsynth für die mRNA-Sequenzierung die unten aufgeführten wichtigsten Ergebnisse:
- Bewertung der Quantität und Qualität der Sequenzierung (im .xlsx-Format)
Bewerten Sie die Quantität und Qualität der Sequenzierungsdaten. - Rohdaten (pro Probe, im .fastq-Format)
Zugriff auf Rohdaten für individuelle Analysen oder als Referenz. - Ein zusammenfassender Projektbericht (.pdf-Format)
Fassen Sie die wichtigsten Projektparameter zusammen.
Mit Bioinformatik
Standard-Bioinformatik-Analyse
Für unsere mRNA-Sequenzierungsanwendung liefert das Microsynth-Modul eine Vielzahl von Erkenntnissen, die auf Ihre wissenschaftlichen Ziele ausgerichtet sind:
Expressionsanalyse:
- Umfassender Bericht (interaktives .html-Format)
Tauchen Sie tiefer in die Daten ein, sortieren und filtern Sie die Ergebnisse interaktiv. - Alignment/Map-Dateien und Indizes (im .bam- und .bai-Format)
Greifen Sie auf Alignment-/Map-Dateien zusammen mit den entsprechenden Indizes zu, um jeden einzelnen Read bis zum Referenzgenom zu verfolgen. - Read Counts zu annotierten Merkmalen (im .tsv-Format)
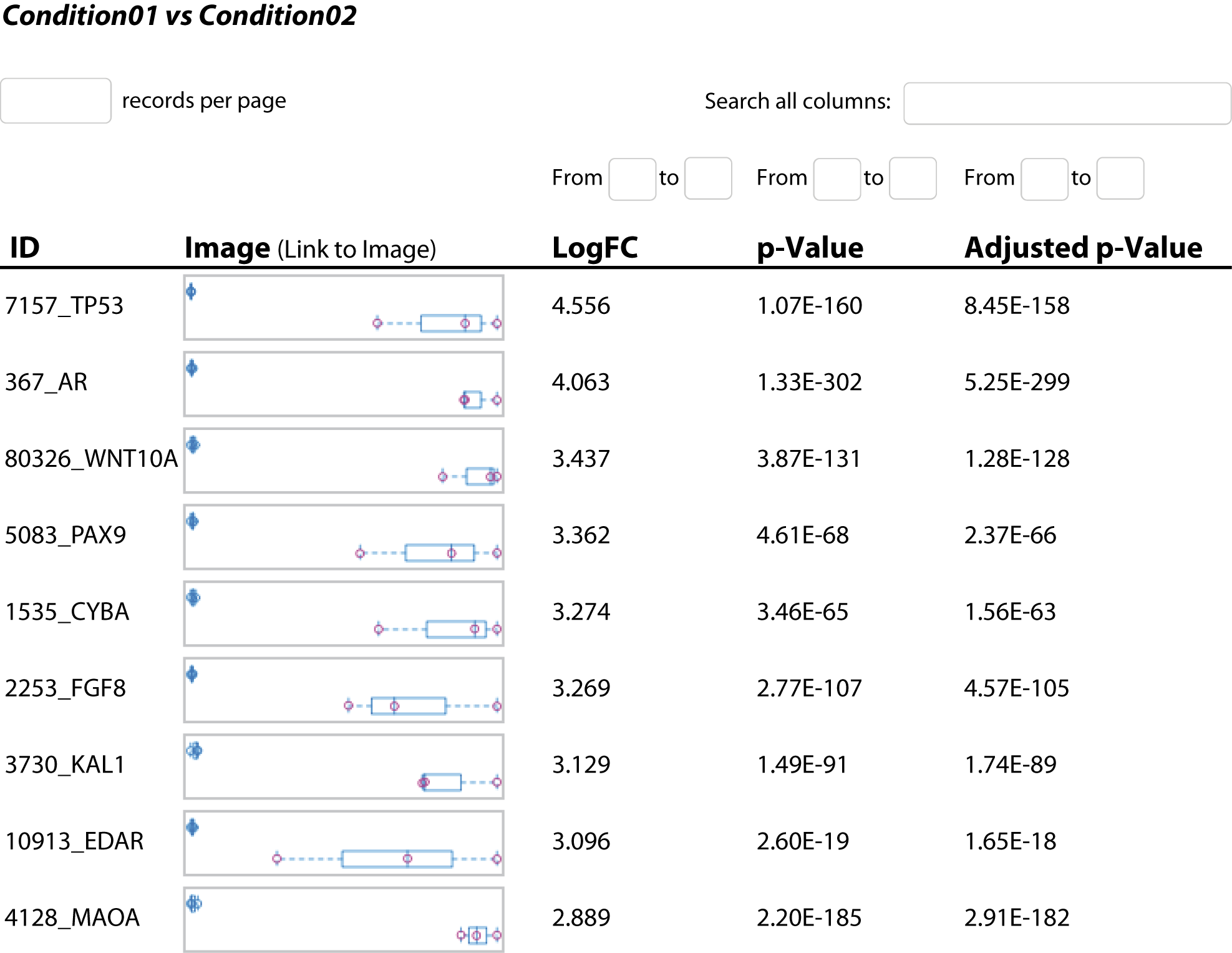
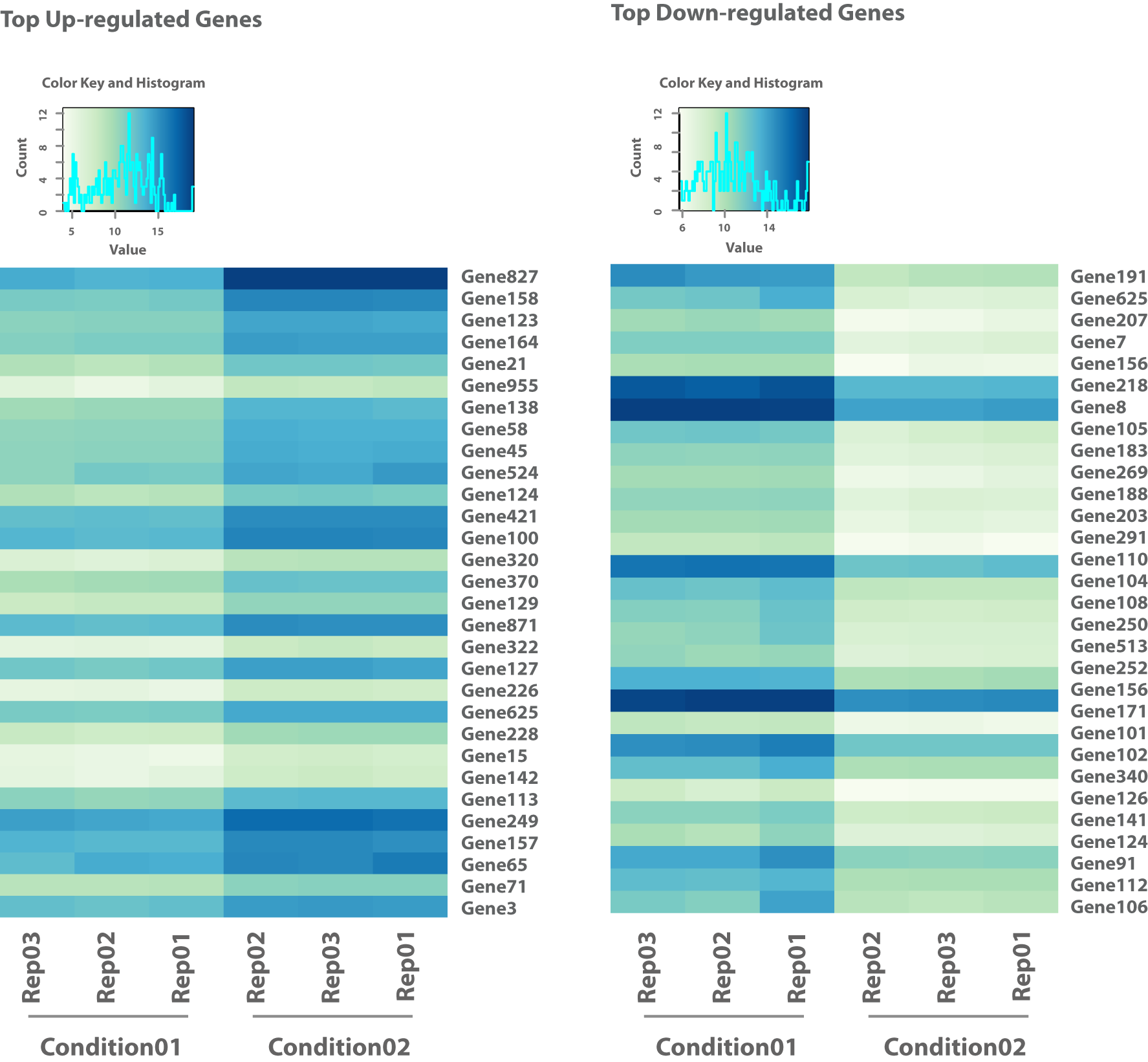
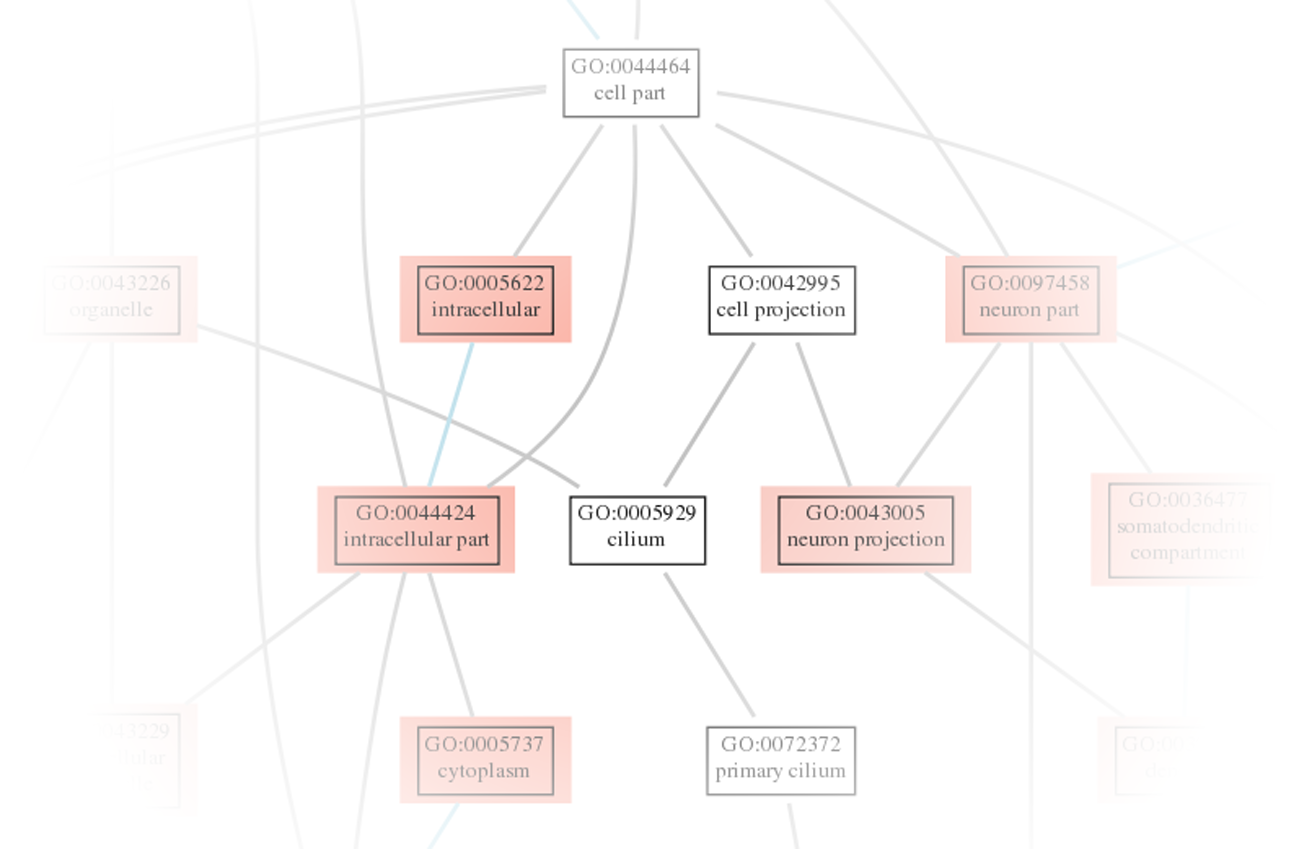
Erhalten Sie Read-Zahlen, die annotierten Merkmalen wie Genen oder Transkripten zugeordnet sind, um eine detaillierte quantitative Analyse durchzuführen. - Visualisierung der statistischen Analyse (z. B. im .png-Format, siehe Abbildung 1-5)
Greifen Sie auf visuell informative Darstellungen von statistischen Analysen zu, einschließlich Vulkandiagrammen, Heatmaps, angereicherten Pfaden und Exon-Karten.
Exklusiv für eukaryotische Transkriptome:
- Differenzielle Genexpressionsanalyse (im .tsv- und interaktiven .html-Format, siehe Abbildung 3)
Erkunden Sie die differentielle Genexpression mit umfassenden Ergebnissen im Tabellen- und HTML-Format. - Geneset-Analyse (im .tsv- und .html-Format, siehe Abbildung 4)
Entdecken Sie angereicherte Pfade durch die Geneset-Analyse, die im Tabellen- und HTML-Format verfügbar ist. - Differenzielle Exon-Nutzungsanalyse (im .tsv- und .html-Format, siehe Abbildung 5)
Erforschen Sie das alternative Spleißen durch die differenzielle Exon-Nutzungsanalyse, deren Ergebnisse sowohl im Tabellen- als auch im HTML-Format zur Verfügung stehen.
Diese Ergebnisse liefern verwertbare Daten aus Ihrer mRNA-Sequenzierungsanalyse und ermöglichen aufschlussreiche Entscheidungen.
Ergänzende bioinformatische Analysen ( gegen Aufpreis)
- Variant Calling von SNVs und kleinen (<50bp) InDels im Transkriptom (im .vcf-Format)
Identifizierung von Einzelnukleotidvarianten (SNVs) und kleinen Insertionen/Deletionen (InDels) im Transkriptom durch Variantenaufrufe im .vcf-Format.
Bei entsprechendem Versuchsaufbau und der Verfügbarkeit relevanter öffentlicher Daten bieten diese zusätzlichen Dienste ein tieferes Verständnis Ihrer Proben.
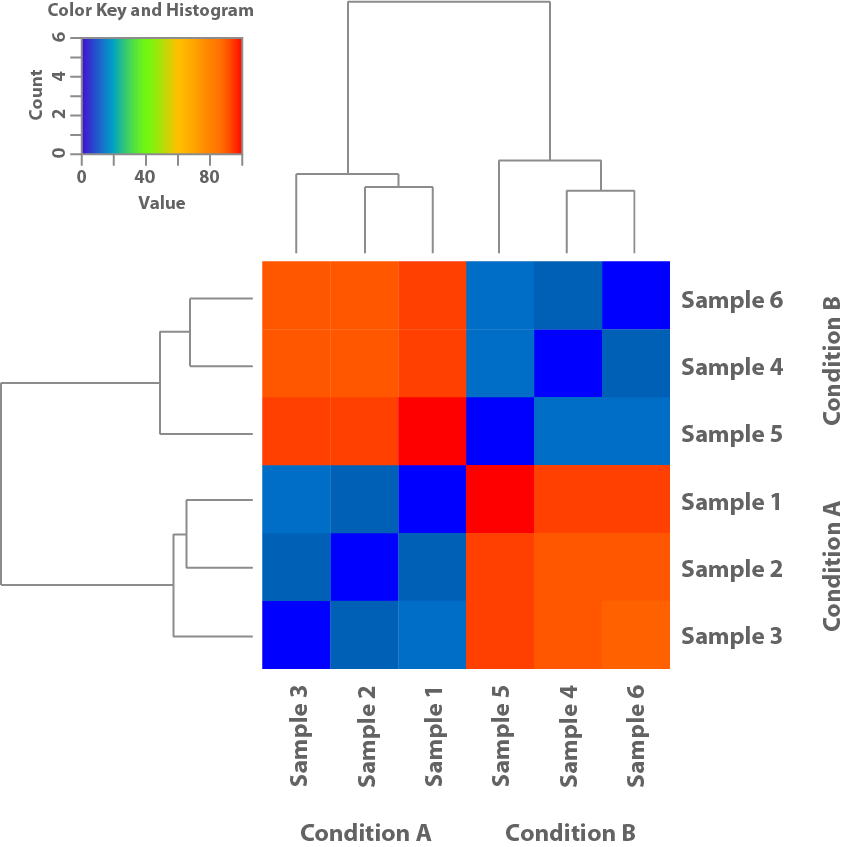
Abbildung 1: Diese Heatmap basiert auf den Expressionsmustern der Proben und zeigt deren Ähnlichkeit zueinander. Dies hilft zu klären, ob die im Experiment verwendeten Bedingungen zu unterschiedlichen Expressionsmustern führen.
Bearbeitungszeiten
- Lieferung der Daten innerhalb von 15 Arbeitstagen nach Probeneingang (einschließlich Library Erstellung und Sequenzierung)
- Zusätzliche 5 Arbeitstage für die RNA Isolation und 3 Arbeitstage für die Datenanalyse (Bioinformatik)
- Express-Service auf Anfrage möglich