Back to top
Amplicon Deep Sequencing

Verwenden Sie Amplicon Deep Sequencing, um:
- eingeführte oder erwartete Mutationen zu erkennen
- seltene genomische Variationen mit hoher Sicherheit zu entdecken
Übersicht
Überlegungen vor dem Start eines Amplicon Deep Sequencing-Projekts:
- Locus-/Amplikonlänge?
- Erwartete Komplexität/Sequenziertiefe?
- DNA-Mengen?
Lassen Sie sich von uns beraten - vom Design bis zur Analyse
Beispielprojekte mit Amplicon Deep Sequencing:
- Taxonomische Analysen mit spezifischem Artenfokus (Barcode of Life Studien)
- CRISPR/Cas und TALEN Verifizierungen und Screenings
- Bestimmung von Arten-Vorkommen (eDNA)
- Alternative zu Hochdurchsatz-Sanger-Sequenzierung für kurze Sequenzen
Anwendungen im Zusammenhang mit der Amplicon Deep Sequencing:
- 16S/ITS Metagenomics
- CRISPR/Cas Sequencing
- Whole Exome Sequencing
Workflow
Ein typischer Workflow für ein Amplikon-Tiefensequenzierungsprojekt ist in der folgenden Grafik dargestellt. Bitte beachten Sie, dass unsere hochmodularen Prozesse Ihnen verschiedene Einstiegs- und Ausstiegsmöglichkeiten bieten. Ob Sie Ihr gesamtes NGS-Projekt an Microsynth auslagern oder nur Teile davon, bleibt Ihnen überlassen.

Weitere Informationen sowie eine detaillierte technische Beschreibung finden Sie in unserer Application Note Amplicon Deep Sequencing (siehe Download auf der rechten Seite).
Resultate
Die von unserem Analysemodul erzeugten Ergebnisse helfen bei der Beantwortung von zwei Hauptfragen der Sequenzierung einer Zielregion (z. B. eines bestimmten Gens; Ausgangsmaterial können Amplikons oder Long Range PCRs sein).
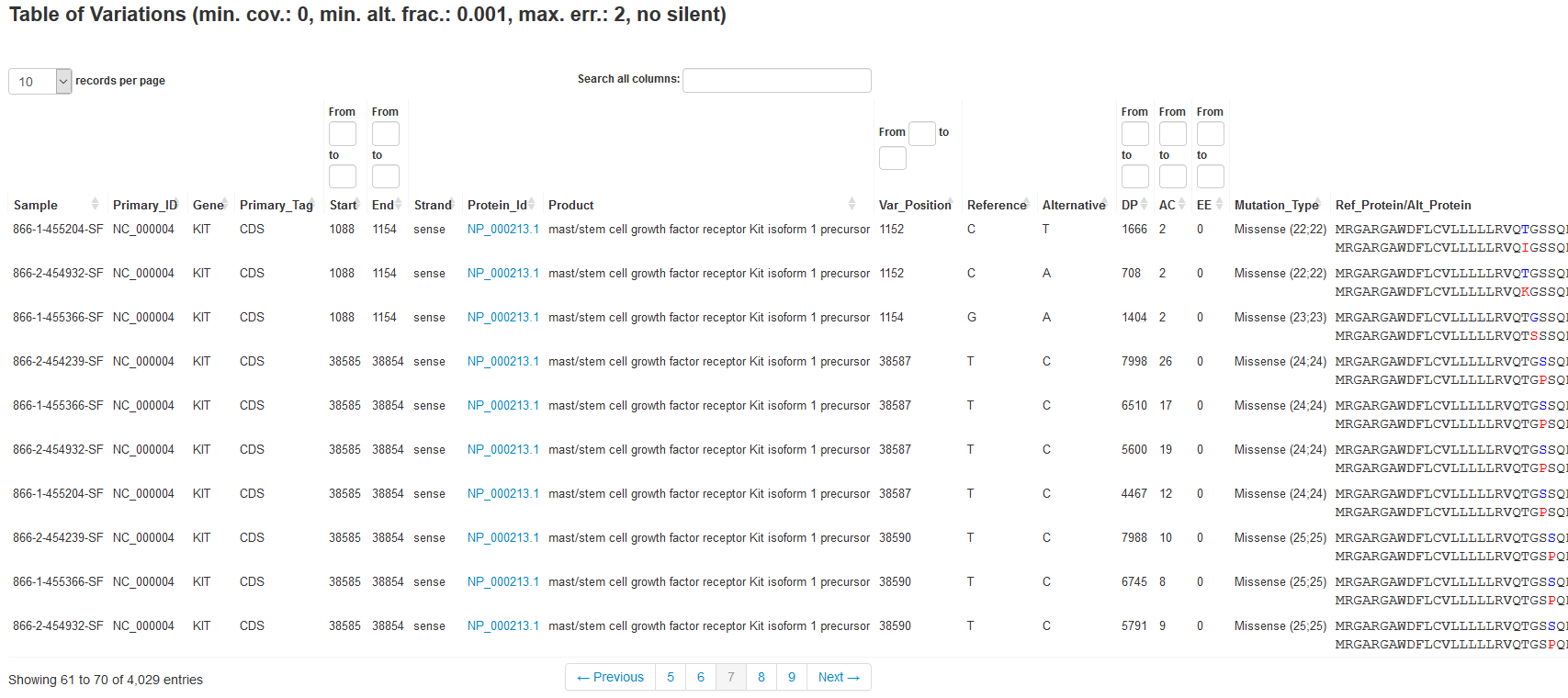
- Welches sind die einzelnen Nukleotidvariationen und kleinen Insertionen/Deletionen im Vergleich zur Referenzsequenz und welche Auswirkungen haben diese gefundenen Variationen auf Proteinebene? (siehe Tabelle 1)
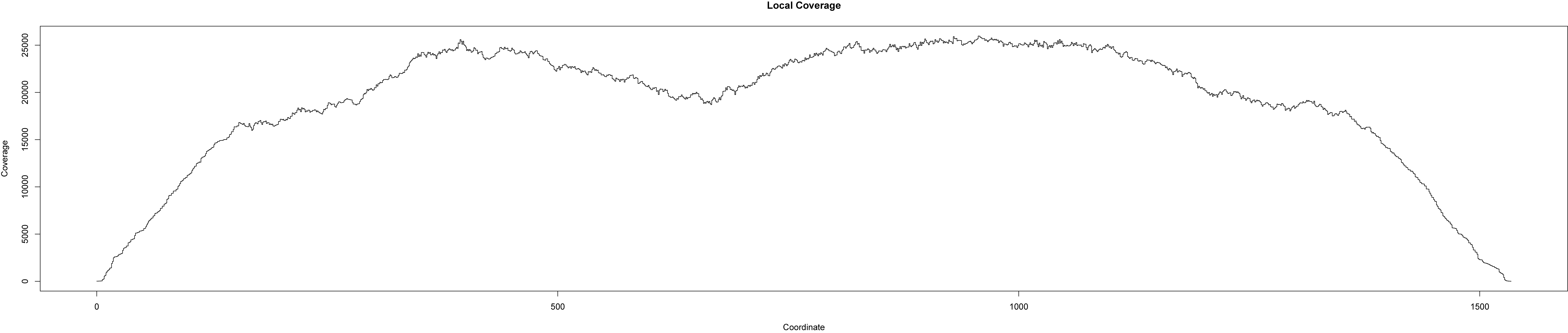
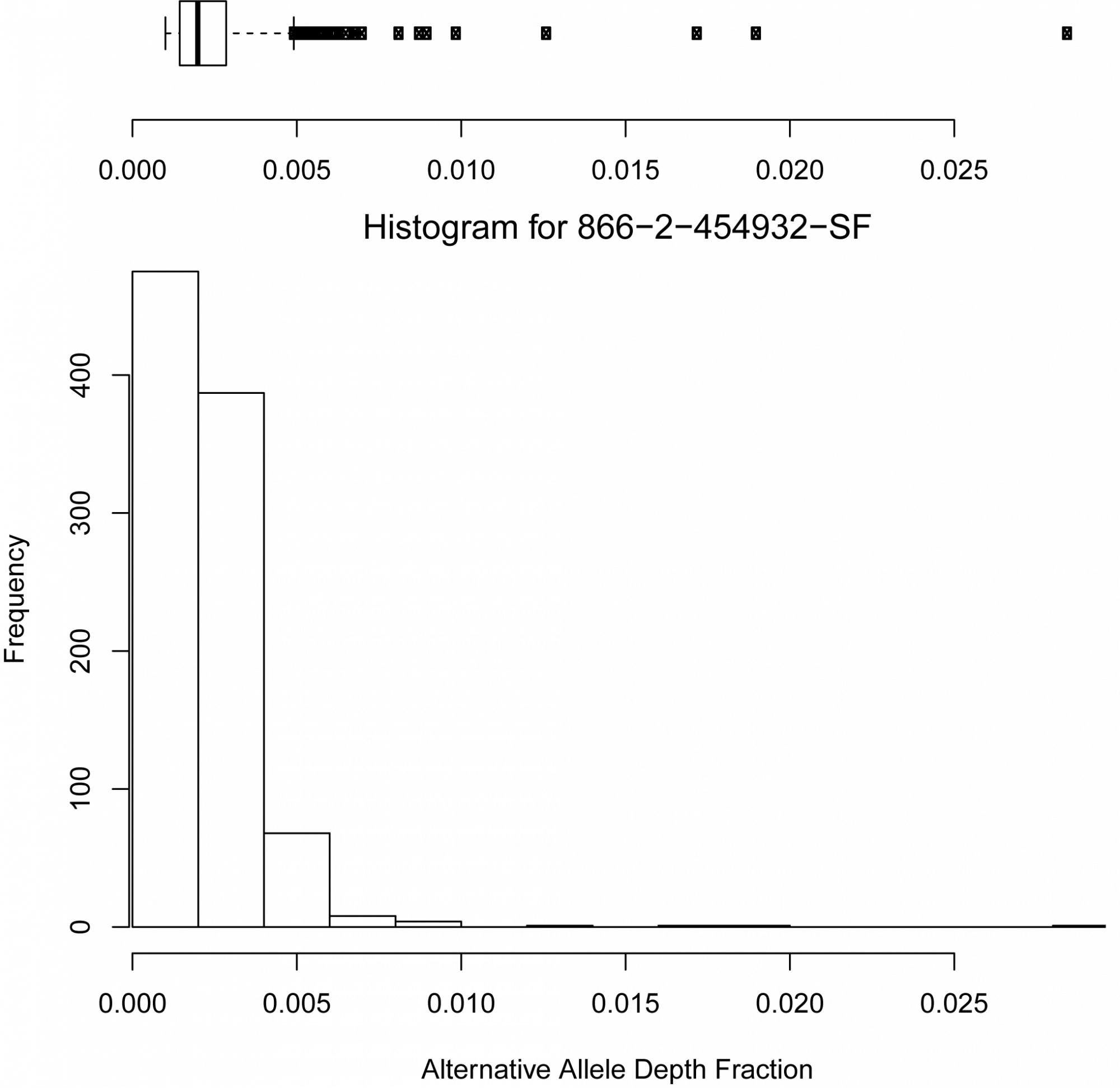
- Wie hoch ist das Vertrauen in die detektierten Variationen und was sind vertrauenswürdige Schwellenwerte, um Rauschen von putativen Variationen zu unterscheiden? (siehe Abbildung 1 und 2)
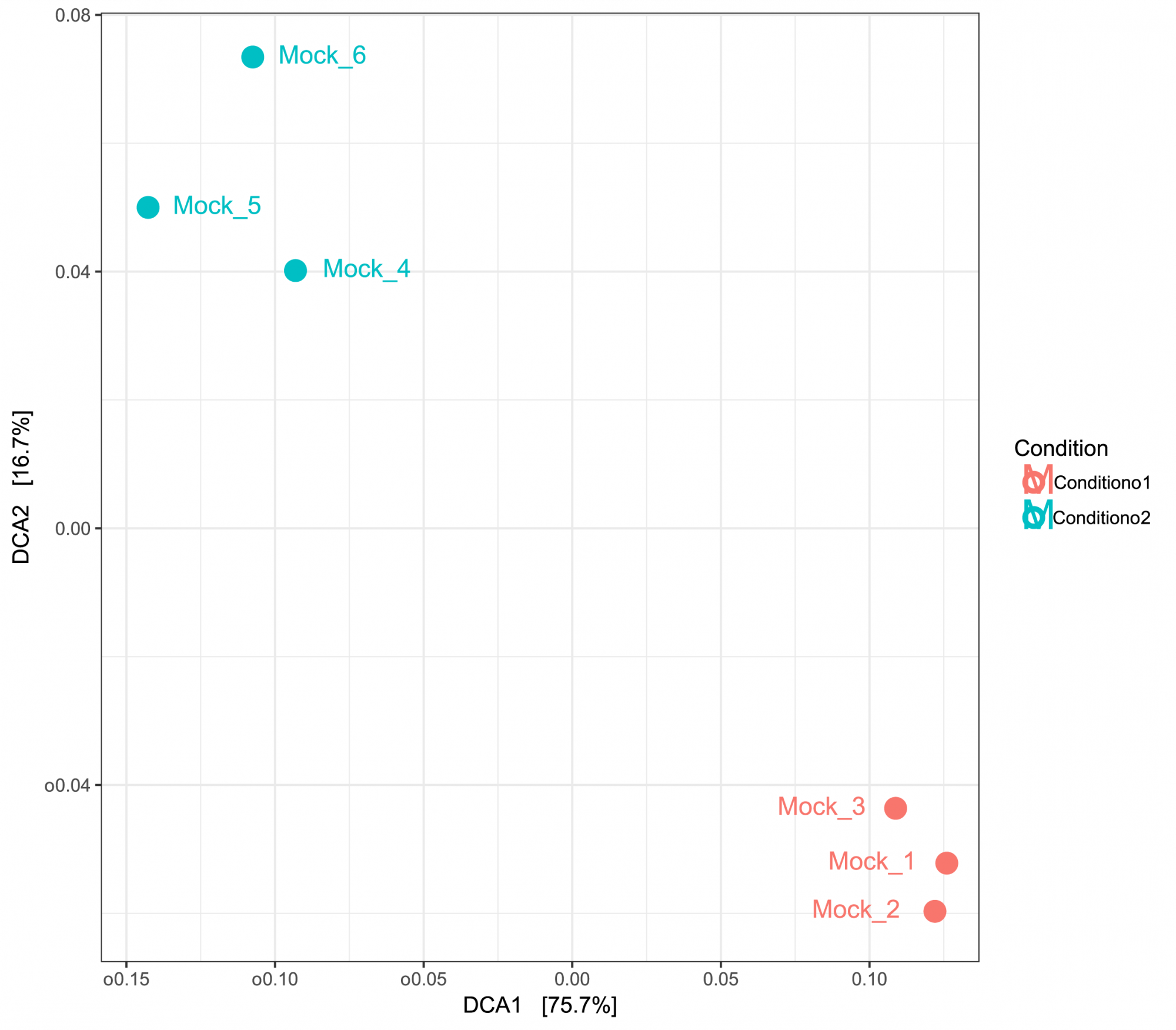
- Was ist die Konsequenz einer bestimmten Behandlung im Vergleich zu einer Kontrollgruppe? (komplementäre vergleichende Dereplikationsanalyse) (siehe Abbildung 3)
Tabelle 1: Dieser Ausschnitt einer Ergebnistabelle listet erkannte Variationen und deren Annotation auf.
Bearbeitungszeiten
- Lieferung der Daten innerhalb von 25 Arbeitstagen nach Probeneingang (inklusive Library Erstellung und Sequenzierung)
- Weitere 10 Arbeitstage für die Datenanalyse (Bioinformatik)
- Express-Service auf Anfrage möglich