Back to top
Whole Exome Sequenzierung

Verwenden Sie die Whole Exome Sequenzierung, um:
- sich auf ausgewählte Regionen und kleine Variationen innerhalb eines Genoms zu konzentrieren
- die Beobachtungen auf die proteincodierenden Regionen eines Genoms in einer diagnostischen Umgebung zu beschränken
- die Kosten und den Zeitaufwand für wiederkehrende Nachweise in einer Langzeitstudie zu reduzieren
Übersicht
Überlegungen vor dem Start eines Whole-Exome-Sequenzierungsprojekts:
- Welcher Organismus und welches Referenzgenom?
- Ziel der Studie?
- Gewünschte Abdeckung?
Lassen Sie sich von uns beraten - vom Design bis zur Analyse
Beispielprojekte mit Whole Exome Sequenzierung:
- Beobachtung von Einzelnukleotid-Variationen (SNVs) und kleinen Insertionen und Deletionen (InDels) bei Krebs-Proben
- Vergleich von SNV-Profilen
- Zeitreihenbeobachtung der Folgen einer Mutagenbehandlung auf die Proteinmodifikation
Anwendungen im Zusammenhang mit der Whole-Exome-Sequenzierung:
- Eukaryotic resequencing
- Microsatellite marker development
Workflow
Ein typischer Workflow für ein Whole-Exome-Sequenzierungsprojekt ist in der folgenden Grafik dargestellt. Bitte beachten Sie, dass unsere hochmodularen Prozesse Ihnen verschiedene Einstiegs- und Ausstiegsmöglichkeiten bieten. Ob Sie Ihr gesamtes NGS-Projekt an Microsynth auslagern oder nur Teile davon, bleibt Ihnen überlassen.
Resultate
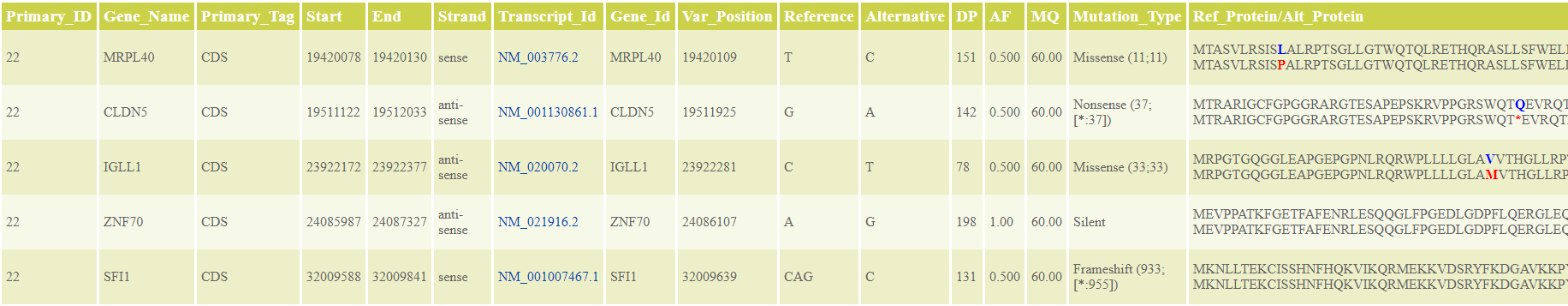
Die von unserem Analysemodul erzeugten Ergebnisse helfen bei der Beantwortung zweier Hauptfragen einer Resequenzierung eines eukaryotischen Exoms mit dem Ziel, Variationen zu einem vorannotierten Referenz-Exom zu erkennen.
- Wie gut wurde das Ziel-Exom abgedeckt? (siehe Tabelle 1)
- Welches sind die einzelnen Nukleotidvariationen und kleinen Insertionen/Deletionen im Vergleich zum Referenzgenom und welche Auswirkungen haben diese gefundenen Variationen auf Proteinebene? (siehe Tabellen 2A und 2B)
- Welche der detektierten Variationen sind auch in öffentlichen Datenbanken (z. B. ClinVar - NCBI - NIH) bekannt und gelistet? (siehe Tabelle 3)
Tabelle 1: Dies ist ein Ausschnitt aus der vollständigen Tabelle mit der Leseabdeckung für jedes Target im sequenzierten Exom.
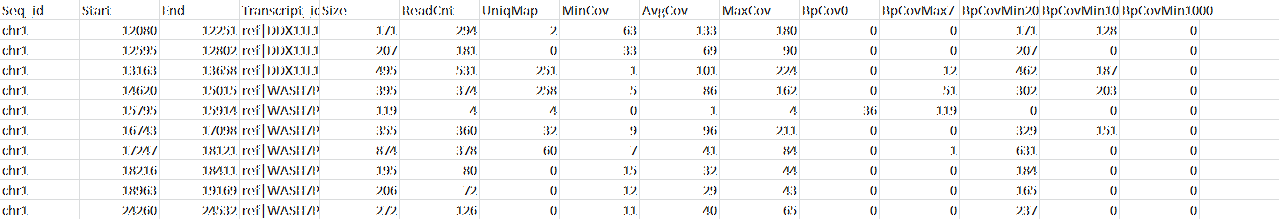
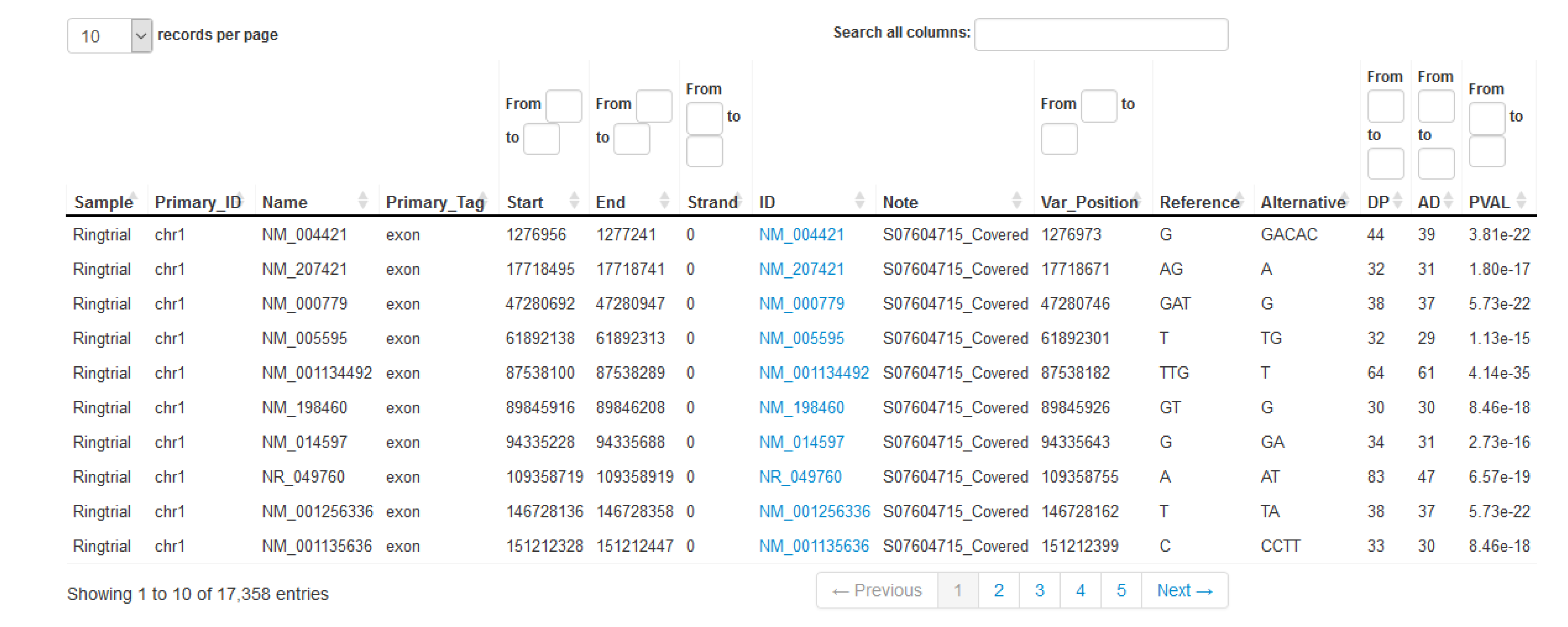
Tabelle 2A: Dieser Ausschnitt aus einer Ergebnistabelle zeigt die erkannten Variationen und deren Beschriftung.
Bearbeitungszeiten
- Lieferung der Daten innerhalb von x Arbeitstagen nach Probeneingang (beinhaltet Library Erstellung und Sequenzierung)
- Zusätzliche x Arbeitstage für die Datenanalyse (Bioinformatik)
- Express-Service auf Anfrage möglich
x= auf Anfrage