Back to top
Kundenspezifische qPCR und dPCR Sonden

Erhöhen Sie die Sensitivität und Spezifität Ihrer real-time oder digitalen PCR mit Sonden von Microsynth. Hochwertige qPCR und dPCR Sonden mit einer breiten Palette an Farbstoffen und Quenchern werden mit ausserorderntlicher Geschwindigkeit geliefert.
Features and Benefits
- Gründliche Aufreinigungsprozesse (HPLC oder sogar PAGE)
- Strenge Qualitätskontrolle (Online-Trityl-Monitoring und MALDI-TOF MS)
- innerhalb von 3 bis 5 Arbeitstagen
Benutzerfreundliches Online-Bestellsystem
-
Einfach zu bedienendes Online-Portal mit einer Reihe von hilfreichen Tools (z.B. Auftragsverfolgung & Historie, komfortable Suche und Nachbestellmöglichkeit)
Umfassendes Leistungsangebot
- Möglichkeit, die Bindungsaffinität über MGB, LNA und andere Tm-Verstärker einzustellen oder sogar verschiedene Tm-Verstärker zu kombinieren, um Ihren Assay individuell zu gestalten
- Große Auswahl an Fluorophoren - Quencher-Kombinationen
- Professioneller Design-Service
- EN ISO 13485:2016 zertifizierter Produktionsprozess
- Die Entwicklung, Validierung, Herstellung und Prüfung von qPCR- und digitalen PCR-Assays kann an Microsynth ausgelagert werden.
- Ausgebildete und erfahrene Wissenschaftler unterstützen Sie gerne
Dual-Labeled Probes
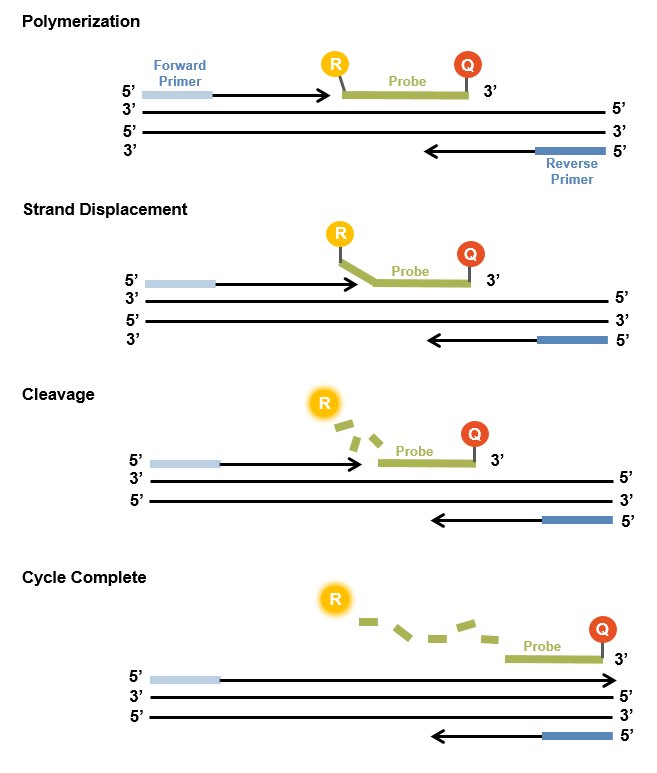
Die sogenannten TaqMan-Sonden sind Hydrolyse-Sonden, die die Spezifität der quantitativen PCR erhöhen sollen.
Im hybridisierten Zustand reicht die Nähe des Fluorophors und des Quenchers aus, um die Fluoreszenz zu quenchen. Während der PCR wird die 5'-3'-Exonukleaseaktivität der Taq-Polymerase genutzt, um die doppelt markierte Sonde zu spalten. Dabei werden der Fluorophor und der Quencher getrennt und ein Fluoreszenzsignal ermöglicht eine quantitative Messung.
Durch die TaqMan-Sonde wird die Spezifität des Nachweises deutlich erhöht .
Wir unterstützen Sie gerne beim Design der Sonden und der dazugehörigen Primer.
| 5‘ Label | 3‘ Label | 0.04 µmol scale1,2,3 | 0.2 µmol scale1,2,3 | 1.0 µmol scale1,2,3 |
|
FAM |
BHQ-1 |
1 OD |
2 OD 10 nmol |
12 OD 60 nmol |
|
AlexaFluor Dyes |
BHQ-1 or BHQ-2 |
0.75 OD 3.75 nmol |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
1 Der Synthesemaßstab bezeichnet die Ausgangsmenge an 3'-Basen (Ausgangsmaterial).
2 Die in OD260 angegebenen Ausbeuten gelten für eine Sonde, die aus 20 DNA-Basen besteht; Berechnung: 1 OD = 5 nmol (bitte beachten Sie, dass diese Berechnung auf Sequenzen mit nahezu homogener Verteilung der 4 DNA-Basen basiert; sie kann bei Sequenzen mit hohem GC-Gehalt >70% etc. abweichen)
3 Die garantierten Ausbeuten für PAGE-gereinigte Oligos sind niedriger.
Double-Quenched Probes
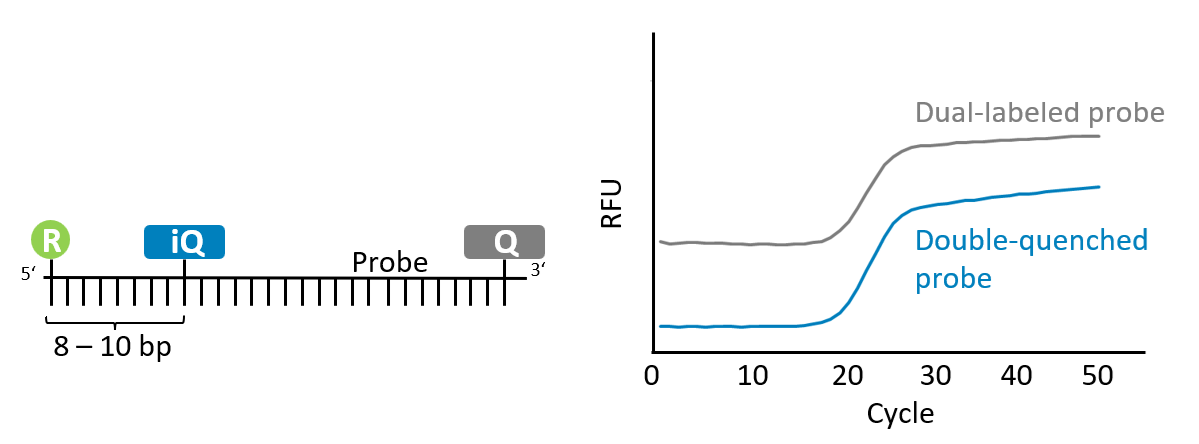
Standard-qPCR-Sonden für 5'-Nuklease-Assays verwenden typischerweise ein Fluorophor am 5'-Ende und einen Quencher am 3'-Ende, um Fluoreszenz zu unterdrücken und die Sondenverlängerung während der PCR zu verhindern. Mit zunehmender Länge der Sonde steigt jedoch oft das Hintergrundsignal, da der Abstand zwischen Fluorophor und Quencher größer wird.
Doppelt-gequenchte Sonden bieten eine überlegene Lösung, indem sie die Hintergrundfluoreszenz reduzieren und die Sensitivität verbessern. Durch die Integration eines zusätzlichen Quenchers 8–10 Basen vom 5'-Fluorophor entfernt wird die Energieübertragung effizienter, was ein deutlich verbessertes Signal-Rausch-Verhältnis ermöglicht. Dieses Design ist besonders vorteilhaft für längere Sonden mit einer Länge von bis zu 40 Nukleotiden.
Unser breites Portfolio an doppelt-gequenchten Sonden
Microsynth bietet eine Vielzahl an doppelt-gequenchten Sonden, die auf Ihre spezifischen Bedürfnisse zugeschnitten sind, einschließlich:
- 5'-FAM-Fluorophor, 3'-BHQ-1-Quencher und interner IQ-500-Quencher für allgemeine Anwendungen.
- Fortschrittliche Fluorophor-Quencher-Kombinationen für anspruchsvolle Anwendungen wie Multiplex-Assays oder herausfordernde Zielsequenzen.
Wir unterstützen Sie gerne bei der Gestaltung der Sonden und der zugehörigen Primer.
| 5‘ Dye | 3‘ Quencher | Internal Quencher | 0.04 µmol scale1,2 | 0.2 µmol scale1,2 | 1.0 µmol scale1,2 |
| FAM | BHQ-1 or TAMRA | iQ500 or iQ530 | 1 OD 5 nmol |
2 OD 10 nmol |
8 OD 40 nmol |
| Yakima Yellow | BHQ-1 | iQ530 | 1 OD 5 nmol |
2 OD 10 nmol |
8 OD 40 nmol |
| HEX | BHQ-2 | iQ530 | 1 OD 5 nmol |
2 OD 10 nmol |
8 OD 40 nmol |
| ROX | BHQ-2 | iQ530 | 0.75 OD 3.75 nmol |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
| CalRed | BHQ-2 | iQ530 | 0.75 OD 3.75 nmol |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
| Cy5 | BHQ-2 | iQ530 | 0.75 OD 3.75 nmol |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
| A647N | BHQ-2 | iQ530 | 0.75 OD 3.75 nmol |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
1 Der Synthesemaßstab bezeichnet die Ausgangsmenge an 3'-Basen (Ausgangsmaterial).
2 Die in OD260 angegebenen Ausbeuten gelten für eine Sonde, die aus 20 DNA-Basen besteht; Berechnung: 1 OD = 5 nmol (bitte beachten Sie, dass diese Berechnung auf Sequenzen mit nahezu homogener Verteilung der 4 DNA-Basen basiert; sie kann bei Sequenzen mit hohem GC-Gehalt >70% etc. abweichen)
Mögliche Anwendungen
- Anspruchsvolle qPCR-Anwendungen mit Bedarf an größerer Flexibilität im Sequenzdesign, z. B. für Targets, die AT-reich sind und längere Sonden (≥25 Basen) erfordern
- ddPCR-Analysen, bei denen die doppelt gequenchte Sonde die Unterscheidung von positiven vs. negativen Tröpfchen erleichtert (verbesserte Clusterbildung)
LNA Probes
Locked Nucleic Acid (LNA) hat sich als leistungsfähiges Werkzeug in vielen qPCR- und digitalen PCR-Anwendungen erwiesen. Wenn sie in DNA-Oligonukleotide eingebaut werden, bieten die resultierenden LNA-haltigen Oligonukleotide folgende Hauptvorteile im Vergleich zu reinen DNA-Basen im nativen Zustand:
- Erhöhte thermische Stabilität und Hybridisierungsspezifität
- Genauere Genquantifizierung und allelische Diskriminierung
- Leichtere und flexiblere Designs für problematische Zielsequenzen
Jede LNA-Base, die in einem Oligo hinzugefügt wird, erhöht die Tm um ca. 2-4 °C. Gesperrte Nukleinsäuresonden können so entworfen werden, dass sie eine ΔTm von >15°C haben. Üblicherweise werden nicht mehr als 5 LNA-Basen in eine Sondensequenz eingebaut. Bitte beachten Sie beim Design und der Optimierung Ihrer LNA-Sonden die hier beschriebenen Richtlinien.
Bei Microsynth können Sie Ihre LNA-Sonde mit der gesamten Palette an Farbstoffen und Quenchern bestellen, die wir auch für unsere dual gelabelten Sonden anbieten.
| 0.04 µmol scale1,2 | 0.2 µmol scale1,2 | 1.0 µmol scale1,2 | ||
| LNA probes |
0.5 OD |
1 OD 5 nmol |
6 OD 30 nmol |
|
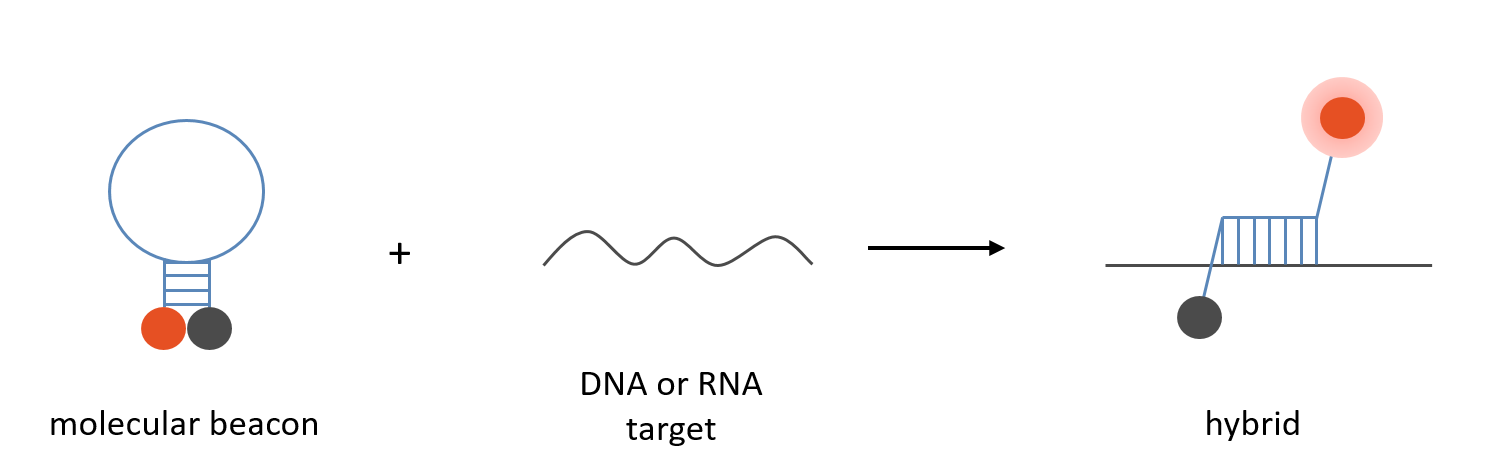
Molecular Beacons
Molecular Beacons werden technisch als doppelt markierte Sonden behandelt. Es gelten die gleichen Spezifikationen und Ausbeuten.
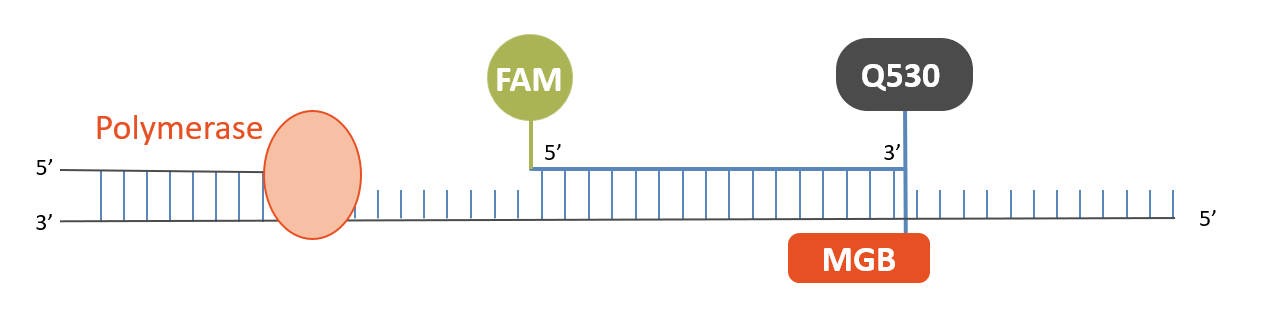
MGB Probes
nbsp]
DNA-Sonden, die mit Minor Groove Binder (MGB) konjugiert sind, bilden außergewöhnlich stabile Duplexe mit einzelsträngigen DNA-Targets, wodurch kürzere Sonden für Hybridisierungs-basierte Assays verwendet werden können.
Daher bietet eine MGB-Sonde folgende Vorteile gegenüber herkömmlichen einfach oder doppelt gequenchten Sonden:
- höhere Selektivität der Zielbindung
- geringere Hintergrundfluoreszenz
- höhere Qualität der Sonde aufgrund der kürzeren Länge
| 5‘ Label | 3‘ Label | 0.04 µmol scale1,2,3 | 0.2 µmol scale1,2,3 | 1.0 µmol scale1,2,3 |
| FAM JOE Yakima Yellow HEX |
MGB-Q5304 |
1 OD
5 nmol
|
2 OD 10 nmol |
10 OD 50 nmol |
1 Der Synthesemassstab entspricht der Ausgangsmenge an 3'-Basen (Ausgangsmaterial).
2 Die in OD260 angegebenen Ausbeuten gelten für eine Sonde, die aus 20 DNA-Basen besteht; Berechnung: 1 OD = 5 nmol (bitte beachten Sie, dass diese Berechnung auf Sequenzen mit nahezu homogener Verteilung der 4 DNA-Basen basiert; sie kann bei Sequenzen mit hohem GC-Gehalt >70% etc. abweichen)
3 Bei MGB-Sonden, die als PAGE bestellt werden, kann die Ausbeute deutlich geringer sein.
4 Darüber hinaus bietet Microsynth den kostengünstigen 3'-Quencher MGB-Q500 an. Dieser Quencher ist nur mit FAM als Reporterfarbstoff erhältlich und kann einen guten Einstieg darstellen. Für höhere Anforderungen (Top-Quenching, Multiplexing) ist 3' MGB-Q530 der Quencher der Wahl.
Wie bestellen
- Besuchen Sie unseren Webshop
- Klicken Sie auf DNA im Bereich "DNA/RNA Synthesis"
- Wählen Sie entweder "Normal Entry", um die gewünschten Sequenzinformationen etc. einzutippen oder zu kopieren/einzufügen, oder wählen Sie alternativ "Upload Entry" unter Verwendung unserer praktischen Excel-Vorlage (kann bei der Bestellung heruntergeladen werden)
- Single-quenched probes (TaqMan, Molecular Beacon, MGB and LNA probes): Wählen Sie die gewünschte 5'- und 3'-Modifikation sowie die HPLC-Aufreinigung1, und Ihr Oligo wird als single-quenched probe erkannt und verarbeitet.
- Bei LNA Sonden fügen Sie die Ordnungssymbole "5, 6, 7 und 8" in Ihre Sequenz ein und wählen unter "Innere Modifikation (5=...)" "LNA-A, LNA-C, LNA-G und LNA-T" usw.
- Double-quenched probes: Fügen Sie das Ordnungssymbol "5" in Ihre Sequenz ein (z.B. catattgaa5actgggttaacggaatt) und wählen Sie unter "Innere Modifikation (5=...") "Internal Quencher 500". Wählen Sie Ihre gewünschte 5'-, 3'-Modifikation sowie die HPLC-Aufreinigung aus, und Ihr Oligo wird als doppelt gequenchte Sonde erkannt und prozessiert.
- Folgen Sie den weiteren Anweisungen
1 Microsynth liefert qPCR-Sonden von höchster Qualität. Der Großteil der Microsynth qPCR-Sonden wird mittels HPLC aufgereinigt, während einige wenige eine PAGE-Aufreinigung benötigen, um die strengen QC-Spezifikationen zu erfüllen. Wenn Ihre Farbstoff/Löscherkombination nicht mit HPLC kompatibel ist, verwenden Sie einfach PAGE.