Back to top
nanoCORE Amplicon Metagenomics (16S/18S/ITS)

Entdecken Sie die Welt mikrobieller Gemeinschaften mit nanoCORE Amplicon Metagenomics, dem innovativen Service von Microsynth basierend auf Oxford Nanopore Sequenzierung. Unser Service ermöglicht eine markerbasierte Vollsequenzierung und liefert außergewöhnlich präzise taxonomische Einblicke bis auf Artniveau. Ob bei der Analyse von Umweltproben, der Überwachung landwirtschaftlicher Böden oder der Erforschung mikrobieller Vielfalt in medizinischen Anwendungen – unser zuverlässiger und effizienter Service unterstützt Ihre Forschung mit höchster Genauigkeit.
Features and Benefits
Schnell
- Lieferung der Ergebnisse innerhalb von 3 Wochen nach Eingang der Probe
Praktisch
- Komplettes Dienstleistungspaket: Von der Probenisolierung bis zur fortgeschrittenen Library-Präparation, Sequenzierung und umfassenden bioinformatischen Analyse.
- Nutzen Sie unsere Dropbox in Ihrer Nähe für den kostenlosen Versand von Proben.
- Beginnen Sie mit Ihrer Analyse mit nur einer Probe!
Kostengünstig
- Ein kostengünstiger Weg zur Erforschung Ihrer mikrobiellen Gemeinschaft.
- Wählen Sie die Phasen aus, die Sie für Ihre Proben beginnen und abschließen möchten, und wir bieten Ihnen äußerst wettbewerbsfähige Pauschalpreise, die auf Ihre spezifischen Bedürfnisse zugeschnitten sind.
Erhöhte Flexibilität
- Keine Größenbeschränkung des Markerlocus
- Sequenzierung von Längenpolymorphismen
- Erhöhte taxonomische Auflösung
Workflow
Der Arbeitsablauf für den nanoCORE Amplicon Metagenomics Service ist in der folgenden Grafik dargestellt. Bitte beachten Sie, dass unser modularer Prozess verschiedene Einstiegsmöglichkeiten bietet.
Resultate
Standard Bioinformatikanalyse
Mit dem Standard-Bioinformatikmodul von Microsynth für nanoCORE Amplicon Metagenomics erhalten Sie umfassende Einblicke:
- Umfassender Bericht (im interaktiven .html-Format)
Dieser detaillierte Bericht führt Sie durch die Daten und ermöglicht es Ihnen, zentrale Ergebnisse interaktiv zu sortieren und zu filtern. - Operational Taxonomic Units (OTUs)
OTUs im .fasta-Format. - OTU-Abundanz, Taxonomie und Vorhersagesicherheit (im .tsv-Format)
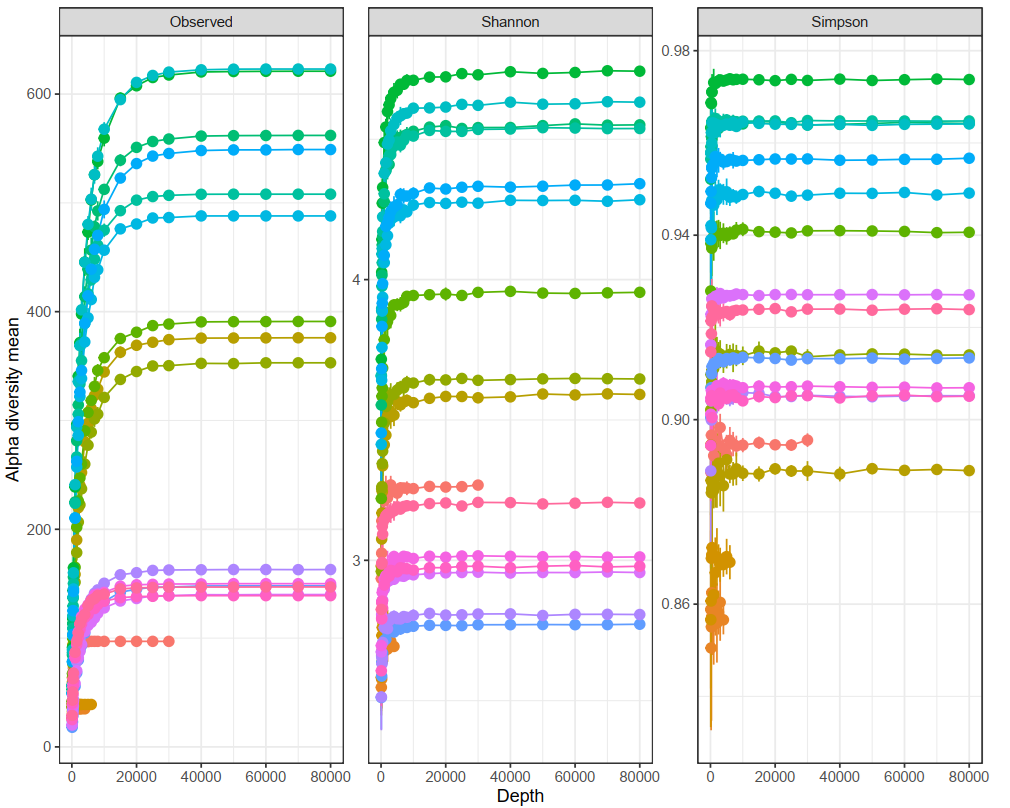
Einblicke in die Abundanz der OTUs, deren taxonomische Zuordnung und die Sicherheit der Vorhersagen. - Analyse der intra- und intersample Diversität und Rarefaktionskurven (im .tsv- und .pdf-Format, siehe Abbildung 1 und 2)
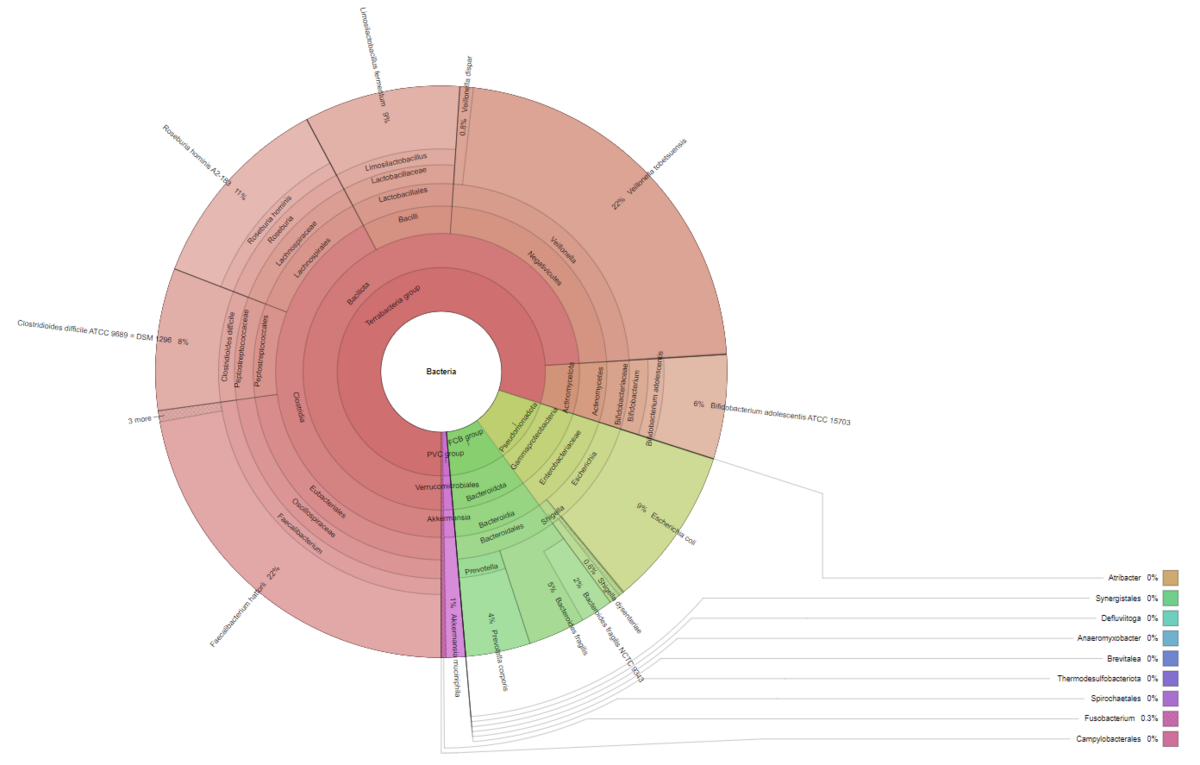
Untersuchen Sie die Diversität innerhalb und zwischen den Proben und schätzen Sie die metagenomische Abdeckung mithilfe von Rarefaktionskurven. - Metagenomische Visualisierung durch Krona-Diagramme (im interaktiven .html-Format, siehe Abbildung 3)
Interpretieren und erkunden Sie Ihre metagenomischen Daten leicht durch interaktive Krona-Diagramme. - Hauptkomponentenanalyse (im .tsv- und .pdf-Format)
Visualisieren und interpretieren Sie komplexe Zusammenhänge mithilfe der Hauptkomponentenanalyse. - Visualisierung der statistischen Analyse
Greifen Sie auf visuell informative Darstellungen der statistischen Analysen zu.
Diese Ergebnisse bieten ein umfassendes Verständnis Ihres Experiments – von der Übersicht bis hin zur nukleotidgenauen Ebene – und befähigen Sie, fundierte Entscheidungen zu treffen und aussagekräftige Schlussfolgerungen zu ziehen.
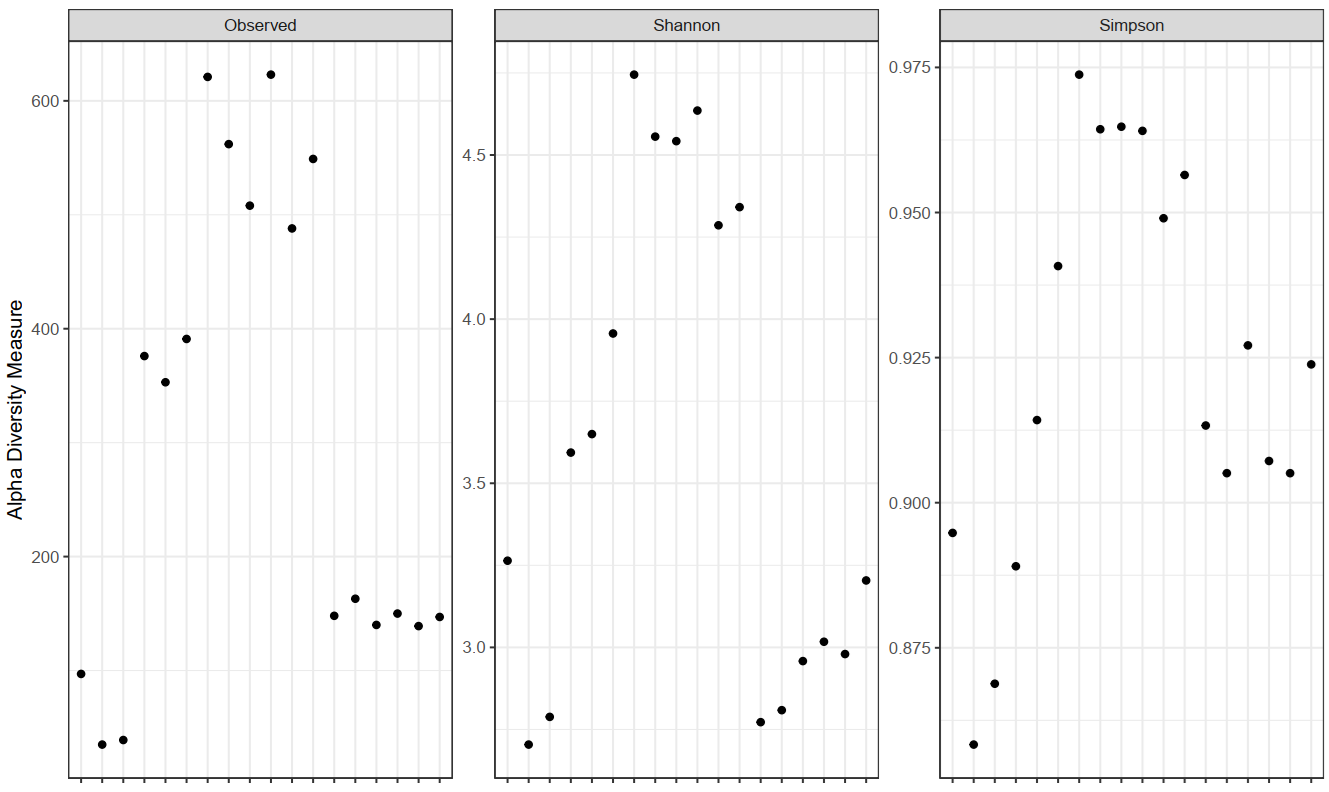
Abbildung 1: Alpha-Diversitätsmasse der analysierten Gemeinschaft, einschließlich beobachteter Artenvielfalt, Simpson-Indizes und Shannon-Diversitätsindizes, die die geschätzte Artenvielfalt und Gleichverteilung darstellen.
Bearbeitungszeiten
- Lieferung der Daten innerhalb von 10 Arbeitstagen nach Probeneingang (einschließlich Vorbereitung der Library, Sequenzierung und Datenanalyse/Bioinformatik).
- Zusätzliche 5 Tage für die DNA-Isolierung